संरक्षित अनुक्रम

का बहु अनुक्रम संरेखण अनुक्रम प्रोटीन के अवशेष 120-180 के लिए अमीनो अम्ल हैं। सभी अनुक्रमों में संरक्षित अवशेषों को ग्रे रंग में हाइलाइट किया गया है। प्रोटीन अनुक्रम संरेखण की प्रत्येक साइट (अर्थात, स्थिति) के नीचे कुंजी है जो संरक्षित साइटों (*), रूढ़िवादी प्रतिस्थापन वाली साइटें (:), अर्ध-रूढ़िवादी प्रतिस्थापन वाली साइटें (।), और गैर-रूढ़िवादी प्रतिस्थापन ( ) को दर्शाती है।[1]
विकासवादी जीव विज्ञान में, संरक्षित अनुक्रम न्यूक्लिक अम्ल (डीएनए और आरएनए) या प्रजातियों में प्रोटीन (ऑर्थोलॉगस अनुक्रम) या जीनोम के अंदर (पैरालॉगस अनुक्रम), या दाता और रिसेप्टर टैक्सा (ज़ेनोलॉगस अनुक्रम) के मध्य समान या समान अनुक्रम होते हैं। संरक्षण प्रदर्शित करता है कि प्राकृतिक चयन द्वारा अनुक्रम बनाए रखा गया है।
अत्यधिक संरक्षित अनुक्रम वह है जो फ़ाइलोजेनेटिक पेड़ से लेकर अब तक भूवैज्ञानिक समय में भी अपेक्षाकृत अपरिवर्तित रहा है। अत्यधिक संरक्षित अनुक्रमों के उदाहरणों में जीवन के सभी क्षेत्रों में उपस्थित राइबोसोम के आरएनए घटक, यूकेरियोट्स में व्यापक होमोबॉक्स अनुक्रम और बैक्टीरिया में टीएमआरएनए सम्मिलित हैं। अनुक्रम संरक्षण का अध्ययन जीनोमिक्स, प्रोटिओमिक्स, विकासवादी जीव विज्ञान, फाइलोजेनेटिक्स, जैव सूचना विज्ञान और गणित के क्षेत्रों के साथ ओवरलैप होता है।
इतिहास
आनुवंशिकता में डीएनए की भूमिका का शोध, और 1949 में फ्रेडरिक सिंगर द्वारा पशु इंसुलिन के मध्य भिन्नता के अवलोकन ने,[2] प्रारंभिक आणविक जीवविज्ञानियों को आणविक दृष्टिकोण से वर्गीकरण का अध्ययन करने के लिए प्रेरित किया।[3][4] 1960 के दशक के अध्ययनों में हीमोग्लोबिन और साइटोक्रोम सी जैसे ज्ञात ऑर्थोलॉगस प्रोटीन के मध्य समानता के लिए को मापने डीएनए संकरण और प्रोटीन क्रॉस-रिएक्टिविटी प्रौद्योगिकी का उपयोग किया गया था।[5][6] 1965 में, एमिल ज़करकांडल और लिनुस पॉलिंग ने आणविक घड़ी की अवधारणा प्रस्तुत की,[7] जिसमें प्रस्ताव दिया गया कि अमीनो अम्ल प्रतिस्थापन की स्थिर दरों का उपयोग दो जीवों के अपसारी विकास के पश्चात से समय का अनुमान लगाने के लिए किया जा सकता है। जबकि प्रारंभिक फ़ाइलोजेनीज़ जीवाश्म रिकॉर्ड से निकटता से युग्मित होते थे, कुछ जीन भिन्न-भिन्न दरों पर विकसित हुए थे, जिससे आणविक विकास के सिद्धांतों का विकास हुआ।[3][4] मार्गरेट डेहॉफ की 1966 में फेरेडॉक्सिन अनुक्रमों की तुलना से ज्ञात हुआ कि प्राकृतिक चयन जीवन के लिए आवश्यक प्रोटीन अनुक्रमों को संरक्षित और अनुकूलित करने का कार्य करता है।[8]
क्रियाविधि
कई पीढ़ियों से, विकासवादी वंश के जीनोम में न्यूक्लिक अम्ल अनुक्रम यादृच्छिक उत्परिवर्तन और विलोप के कारण समय के साथ धीरे-धीरे परिवर्तित हो सकते हैं।[9][10] क्रोमोसोमल पुनर्व्यवस्था के कारण अनुक्रम भी पुनः संयोजित या या नष्ट हो सकते हैं। संरक्षित अनुक्रम वे अनुक्रम हैं जो ऐसी शक्तियों के अतिरिक्त जीनोम में बने रहते हैं, और पृष्ठभूमि उत्परिवर्तन दर की तुलना में उत्परिवर्तन की धीमी दर होती है।[11]
कोडिंग और गैर-कोडिंग न्यूक्लिक अम्ल अनुक्रमों में संरक्षण हो सकता है। माना जाता है कि अत्यधिक संरक्षित डीएनए अनुक्रमों को कार्यात्मक महत्व होता है, चूँकि कई उच्च संरक्षित गैर-कोडिंग डीएनए अनुक्रमों की भूमिका को कम समझा जाता है।[12][13] किसी अनुक्रम को किस सीमा तक संरक्षित किया जाता है, यह भिन्न-भिन्न विकासवादी दबाव, उत्परिवर्तन के लिए इसकी मजबूती, जनसंख्या आनुवंशिकी और आनुवंशिक बहाव से प्रभावित हो सकता है। कई कार्यात्मक अनुक्रम भी मॉड्यूलर होते हैं, जिसमें ऐसे क्षेत्र सम्मिलित हैं जो स्वतंत्र विकासवादी दबाव के अधीन हो सकते हैं, जैसे कि प्रोटीन डोमेन हैं।[14]
कोडिंग अनुक्रम
कोडिंग अनुक्रमों में, न्यूक्लिक अम्ल और अमीनो अम्ल अनुक्रम को भिन्न-भिन्न विस्तार तक संरक्षित किया जा सकता है, क्योंकि आनुवंशिक कोड की विकृति का अर्थ है कि कोडिंग अनुक्रम में समानार्थी प्रतिस्थापन इसके प्रोटीन उत्पाद के अमीनो अम्ल अनुक्रम को प्रभावित नहीं करता है।[15]
प्रोटीन या डोमेन की संरचना या कार्य को बनाए रखने के लिए अमीनो अम्ल अनुक्रमों को संरक्षित किया जा सकता है। संरक्षित प्रोटीन अल्प अमीनो अम्ल प्रतिस्थापन से निकलते हैं, या समान जैव रासायनिक गुणों वाले अमीनो अम्ल को प्रतिस्थापित करने की संभावना अधिक होती है।[16] अनुक्रम के अंदर, अमीनो अम्ल जो फोल्डिंग, संरचनात्मक स्थिरता के लिए महत्वपूर्ण हैं, या जो बाध्यकारी साइट बनाते हैं, अधिक उच्च संरक्षित हो सकते हैं।[17][18]
प्रोटीन कोडिंग जीन के न्यूक्लिक अम्ल अनुक्रम को अन्य चयनात्मक दबावों द्वारा भी संरक्षित किया जा सकता है। कुछ जीवों में कोडन उपयोग पूर्वाग्रह अनुक्रम में समानार्थक उत्परिवर्तन के प्रकारों को प्रतिबंधित कर सकता है। कोडिंग जीन के एमआरएनए में द्वितीयक संरचना का कारण बनने वाले न्यूक्लिक अम्ल अनुक्रमों का चयन किया जा सकता है, क्योंकि कुछ संरचनाएं अनुवाद को नकारात्मक रूप से प्रभावित कर सकती हैं, या संरक्षित हो सकती हैं जहां एमआरएनए कार्यात्मक गैर-कोडिंग आरएनए के रूप में भी कार्य करता है।[19][20]
गैर-कोडिंग
जीन विनियमन के लिए महत्वपूर्ण गैर-कोडिंग अनुक्रम, जैसे राइबोसोम और प्रतिलेखन कारकों के बंधन या मान्यता स्थल, को जीनोम के अंदर संरक्षित किया जा सकता है। उदाहरण के लिए, संरक्षित जीन या ऑपेरॉन के प्रवर्तक को भी संरक्षित किया जा सकता है। प्रोटीन के जैसे, न्यूक्लिक अम्ल जो गैर-कोडिंग आरएनए (एनसीआरएनए) की संरचना और कार्य के लिए महत्वपूर्ण हैं, चूँकि, प्रोटीन-कोडिंग अनुक्रमों की तुलना में एनसीआरएनए में अनुक्रम संरक्षण सामान्यतः खराब होता है, और संरचना या कार्य में योगदान देने वाले आधार जोड़े को प्रायः इसके अतिरिक्त संरक्षित होते हैं।[21][22]
पहचान
संरक्षित अनुक्रमों को सामान्यतः अनुक्रम संरेखण के आधार पर जैव सूचना विज्ञान दृष्टिकोणों द्वारा पहचाना जाता है। उच्च-थ्रूपुट डीएनए अनुक्रमण और प्रोटीन मास स्पेक्ट्रोमेट्री ने 2000 के दशक के प्रारंभ से तुलना के लिए प्रोटीन अनुक्रमों और संपूर्ण जीनोम की उपलब्धता में अधिक वृद्धि की है।[23][24]
समरूपता शोध
ब्लास्ट, एचएमएमईआर, ऑर्थोलॉगआर और इनफर्नल जैसे उपकरणों का उपयोग करते हुए, होमोलॉजी शोध द्वारा संरक्षित अनुक्रमों की पहचान की जा सकती है।[25][26] होमोलॉजी शोध उपकरण इनपुट के रूप में व्यक्तिगत न्यूक्लिक अम्ल या प्रोटीन अनुक्रम को ले सकते हैं, या ज्ञात संबंधित अनुक्रमों के कई अनुक्रम संरेखण से उत्पन्न सांख्यिकीय मॉडल का उपयोग कर सकते हैं। सांख्यिकीय मॉडल जैसे प्रोफाइल-एचएमएम, और आरएनए सहप्रसरण मॉडल जो संरचनात्मक सूचना भी सम्मिलित करते हैं,[27] अधिक दूर से संबंधित अनुक्रमों का शोध करते समय सहायक हो सकते हैं। तब इनपुट अनुक्रमों को संबंधित व्यक्तियों या अन्य प्रजातियों के अनुक्रमों के डेटाबेस के विरुद्ध संरेखित किया जाता है। परिणामी संरेखण को युग्मित करने वाले अमीनो अम्ल या आधारों की संख्या और संरेखण द्वारा उत्पन्न अंतराल या विलोपन की संख्या के आधार पर स्कोर किया जाता है। स्वीकार्य रूढ़िवादी प्रतिस्थापन की पहचान PAM और BLOSUM जैसे प्रतिस्थापन मेट्रिसेस का उपयोग करके की जा सकती है। उच्च स्कोरिंग संरेखण को सजातीय अनुक्रमों से माना जाता है। अनुक्रम के संरक्षण को व्यापक फ़िलेजेनेटिक रेंज में अत्यधिक समान समरूपों को ज्ञात करके किया जा सकता है।[28]
एकाधिक अनुक्रम संरेखण
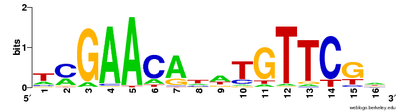
संरक्षित अनुक्रमों को देखने के लिए एकाधिक अनुक्रम संरेखण का उपयोग किया जा सकता है। क्लस्टल प्रारूप में संरेखण के संरक्षित स्तंभों को एनोटेट करने के लिए सादा-पाठ कुंजी सम्मिलित है, जो संरक्षित अनुक्रम (*), रूढ़िवादी उत्परिवर्तन (:), अर्ध-रूढ़िवादी उत्परिवर्तन (।), और गैर-रूढ़िवादी उत्परिवर्तन ( ) को दर्शाता है।[30] अनुक्रम लोगो ऊंचाई से संरेखण में प्रत्येक बिंदु पर वर्णों के अनुपात का प्रतिनिधित्व करके संरक्षित अनुक्रम भी दिखा सकते हैं।[29]
जीनोम संरेखण
संपूर्ण जीनोम संरेखण (डब्ल्यूजीए) का उपयोग प्रजातियों में अत्यधिक संरक्षित क्षेत्रों की पहचान करने के लिए भी किया जा सकता है। वर्तमान में पुनर्व्यवस्था, दोहराए जाने वाले क्षेत्रों और कई यूकेरियोटिक जीनोम के बड़े आकार से निपटने की कम्प्यूटेशनल जटिलता के कारण डब्ल्यूजीए उपकरणों की त्रुटिहीनता और मापनीयता सीमित है।[32] चूँकि, 30 या अधिक निकट संबंधी जीवाणुओं (प्रोकैरियोट्स) के डब्ल्यूजीए अब तीव्रता से संभव हो रहे हैं।[33][34]
स्कोरिंग प्रणाली
अन्य दृष्टिकोण सांख्यिकीय परीक्षणों के आधार पर संरक्षण के मापन का उपयोग करते हैं जो उन अनुक्रमों की पहचान करने का प्रयास करते हैं जो अपेक्षित पृष्ठभूमि (तटस्थ) उत्परिवर्तन दर से भिन्न रूप से उत्परिवर्तित होते हैं।
जीईआरपी (जीनोमिक इवोल्यूशनरी रेट प्रोफाइलिंग) रूपरेखा प्रजातियों में आनुवंशिक अनुक्रमों का संरक्षण करती है। यह दृष्टिकोण कई अनुक्रम संरेखण से प्रजातियों के समूह में तटस्थ उत्परिवर्तन की दर का अनुमान लगाता है, और फिर अनुक्रम के उन क्षेत्रों की पहचान करता है जो अपेक्षा से कम उत्परिवर्तन प्रदर्शित करते हैं। इन क्षेत्रों को तब देखी गई उत्परिवर्तन दर और अपेक्षित पृष्ठभूमि उत्परिवर्तन दर के मध्य अंतर के आधार पर अंक दिए जाते हैं। उच्च जीईआरपी स्कोर तब अत्यधिक संरक्षित अनुक्रम को प्रदर्शित करता है।[35][36]
सूची[37][38] (लोकल आइडेंटिटी एंड शेयर्ड टैक्सा) इस धारणा पर आधारित है कि दूर से संबंधित प्रजातियों की तुलना में संरक्षण का आकलन करते समय मानव से निकटता से संबंधित प्रजातियों में देखी गई विविधताएं अधिक महत्वपूर्ण हैं। इस प्रकार, सूची कई अनुक्रम संरेखण (एमएसए) में प्रासंगिक अनुक्रमों की पहचान करने के लिए प्रत्येक स्थिति के निकट स्थानीय संरेखण पहचान का उपयोग करता है और फिर यह मानव के लिए इन अनुक्रमों की वर्गीकरण दूरी के आधार पर संरक्षण का अनुमान लगाता है। अन्य उपकरणों के विपरीत, सूची एमएसए में विविधताओं की गिनती/आवृत्ति पर ध्यान नहीं देता है।
अमीनोड[39] सजातीय प्रोटीन में परिवर्तन का विश्लेषण करने के लिए फ़ाइलोजेनेटिक विश्लेषण के साथ कई संरेखण को जोड़ता है और प्लॉट का उत्पादन करता है जो विकासवादी परिवर्तनों की स्थानीय दरों को प्रदर्शित करता है। यह दृष्टिकोण प्रोटीन में क्रमिक रूप से बाधित क्षेत्रों की पहचान करता है, जो ऐसे खंड हैं जो चयन को शुद्ध करने के अधीन हैं और सामान्यतः सामान्य प्रोटीन कार्य के लिए महत्वपूर्ण हैं।
PhyloP और PhyloHMM जैसे अन्य दृष्टिकोण प्रतिस्थापन दरों की संभाव्यता वितरण की तुलना करने के लिए सांख्यिकीय फाइलोजेनेटिक विधियों को सम्मिलित करते हैं, जो संरक्षण और त्वरित उत्परिवर्तन दोनों को ज्ञात करने की अनुमति देता है। सर्वप्रथम, फ़ाइलोजेनेटिक पेड़ के आधार पर, एकाधिक अनुक्रम संरेखण में एक कॉलम के लिए होने वाले अपेक्षित प्रतिस्थापनों की संख्या से पृष्ठभूमि संभाव्यता वितरण उत्पन्न होता है। रुचि की प्रजातियों के मध्य अनुमानित विकासवादी संबंधों का उपयोग किसी भी प्रतिस्थापन के महत्व की गणना करने के लिए किया जाता है (अर्थात दो निकट संबंधी प्रजातियों के मध्य प्रतिस्थापन दूर से संबंधित लोगों की तुलना में कम होने की संभावना हो सकती है, और इसलिए अधिक महत्वपूर्ण है)। संरक्षण को ज्ञात करने के लिए, एकाधिक अनुक्रम संरेखण के उपसमूह के लिए संभाव्यता वितरण की गणना की जाती है, संभावना-अनुपात परीक्षण या स्कोर परीक्षण जैसे सांख्यिकीय परीक्षण का उपयोग करके पृष्ठभूमि वितरण की तुलना की जाती है। दो वितरणों की तुलना करने से उत्पन्न P-मानों का उपयोग संरक्षित क्षेत्रों की पहचान करने के लिए किया जाता है। PhyloHMM संभाव्यता वितरण उत्पन्न करने के लिए छिपे हुए मार्कोव मॉडल का उपयोग करता है। PhyloP सॉफ़्टवेयर पैकेज संभावना-अनुपात परीक्षण या स्कोर परीक्षण के साथ-साथ जीईआरपी-जैसी स्कोरिंग प्रणाली का उपयोग करके संभाव्यता वितरण की तुलना करता है।[40][41][42]
अत्यधिक संरक्षण
अति-संरक्षित तत्व
अति-संरक्षित तत्व या यूसीई ऐसे अनुक्रम हैं जो कई वर्गीकरण समूहों में अत्यधिक समान या समान हैं। इनका सर्वप्रथम कशेरुकियों जंतुओं में शोध किया गया था,[43] और पश्चात में व्यापक रूप से भिन्न टैक्सा के अंदर पहचाने गए हैं।[44] जबकि यूसीई की उत्पत्ति और कार्य को कम समझा गया है,[45] उनका उपयोग एमनियोट्स, कीड़ों, और जानवरों और पौधों के मध्य गहरे समय के विचलन का परीक्षण करने के लिए किया गया है।[46] [47][48]
सार्वभौमिक रूप से संरक्षित जीन
सबसे अधिक संरक्षित जीन वे हैं जो सभी जीवों में पाए जा सकते हैं। इनमें मुख्य रूप से प्रतिलेखन और अनुवाद के लिए आवश्यक एनसीआरएनए और प्रोटीन सम्मिलित हैं, जिन्हें सभी जीवन के अंतिम सार्वभौमिक सामान्य पूर्वज से संरक्षित माना जाता है।[49]
जिन जीनों या जीन परिवारों को सार्वभौमिक रूप से संरक्षित पाया गया है उनमें जीटीपी-बाध्यकारी बढ़ाव कारक परिवार, मेथियोनीन एमिनोपेप्टिडेज़ 2, सेरीन हाइड्रोक्सीमिथाइलट्रांसफेरेज़ और एटीपी ट्रांसपोर्टर सम्मिलित हैं।[50] ट्रांसक्रिप्शन मशीनरी के घटक, जैसे कि आरएनए पोलीमरेज़ और हेलिकेज़, और अनुवाद मशीनरी, जैसे राइबोसोमल आरएनए, टीआरएनए और राइबोसोमल प्रोटीन भी सार्वभौमिक रूप से संरक्षित हैं।[51]
अनुप्रयोग
फ़ाइलोजेनेटिक्स और वर्गीकरण
संरक्षित अनुक्रमों के समूह का उपयोग प्रायः फाइलोजेनेटिक पेड़ों को उत्पन्न करने के लिए किया जाता है, क्योंकि यह माना जा सकता है कि समान अनुक्रम वाले जीव निकट से संबंधित हैं।[52] अध्ययन के वर्गीकरण सीमा के आधार पर अनुक्रमों का चयन भिन्न हो सकता है। उदाहरण के लिए, सबसे उच्च संरक्षित जीन जैसे कि 16एस आरएनए और अन्य राइबोसोमल अनुक्रम, गहरे फ़ाइलोजेनेटिक संबंधों के पुनर्निर्माण और मेटागेनोमिक्स अध्ययन में बैक्टीरियल फ़ाइला की पहचान करने के लिए उपयोगी होते हैं।[53][54] अनुक्रम जो क्लेड के अंदर संरक्षित होते हैं किन्तु कुछ उत्परिवर्तन से निकलते हैं, जैसे हाउसकीपिंग जीन, का उपयोग प्रजातियों के संबंधों का अध्ययन करने के लिए किया जा सकता है।[55][56][57] आंतरिक ट्रांसक्राइब्ड स्पेसर (आईटीएस) क्षेत्र, जो संरक्षित आरआरएनए जीनों के मध्य की दूरी के लिए आवश्यक है, किन्तु तीव्रता से विकसित होता है, सामान्यतः कवक और तीव्रता से विकसित होने वाले बैक्टीरिया के उपभेदों को वर्गीकृत करने के लिए उपयोग किया जाता है।[58][59][60][61]
चिकित्सा अनुसंधान
चूंकि अत्यधिक संरक्षित अनुक्रमों में प्रायः महत्वपूर्ण जैविक कार्य होते हैं, वे आनुवंशिक रोगों के कारण की पहचान करने के लिए प्रारंभिक बिंदु के रूप में उपयोगी हो सकते हैं। कई जन्मजात चयापचय संबंधी विकार और लाइसोसोमल भंडारण रोग व्यक्तिगत संरक्षित जीन में परिवर्तन का परिणाम होते हैं, जिसके परिणामस्वरूप विलुप्त या दोषपूर्ण एंजाइम होते हैं जो रोग के लक्षणों के अंतर्निहित कारण होते हैं। आनुवंशिक रोगों की भविष्यवाणी मनुष्यों और चूहों या फल मक्खियों जैसे प्रयोगशाला जीवों के मध्य संरक्षित अनुक्रमों की पहचान करके, और इन जीनों के नॉकआउट के प्रभावों का अध्ययन करके की जा सकती है।[62][63][64] जीनोम-वाइड एसोसिएशन अध्ययन का उपयोग बीमारी या स्वास्थ्य परिणामों से जुड़े संरक्षित अनुक्रमों में भिन्नता की पहचान करने के लिए भी किया जा सकता है। अल्जाइमर रोग में दो दर्जन से अधिक नवीन संभावित संवेदनशीलता लोकी का शोध किया गया था।[65][66]
कार्यात्मक एनोटेशन
संरक्षित अनुक्रमों की पहचान का उपयोग जीन जैसे कार्यात्मक अनुक्रमों के शोध और भविष्यवाणी करने के लिए किया जा सकता है।[67] किसी ज्ञात कार्य के साथ संरक्षित अनुक्रम, जैसे कि प्रोटीन डोमेन, का उपयोग किसी अनुक्रम के कार्य की भविष्यवाणी करने के लिए भी किया जा सकता है। Pfam और संरक्षित डोमेन डेटाबेस जैसे संरक्षित प्रोटीन डोमेन के डेटाबेस का उपयोग पूर्वानुमानित प्रोटीन कोडिंग जीन में कार्यात्मक डोमेन को एनोटेट करने के लिए किया जा सकता है।[68]
यह भी देखें
- विकासवादी विकास जीव विज्ञान
- एनएपीपी (डेटाबेस)
- पृथक्करण स्थल
- अनुक्रम संरेखण
- अनुक्रम संरेखण सॉफ्टवेयर
- आधार
- अति-संरक्षित तत्व
संदर्भ
- ↑ "क्लस्टल एफएक्यू #प्रतीक". Clustal. Archived from the original on 24 October 2016. Retrieved 8 December 2014.
- ↑ Sanger, F. (24 September 1949). "इंसुलिन में प्रजाति अंतर". Nature. 164 (4169): 529. Bibcode:1949Natur.164..529S. doi:10.1038/164529a0. PMID 18141620. S2CID 4067991.
- ↑ 3.0 3.1 Marmur, J; Falkow, S; Mandel, M (October 1963). "बैक्टीरियल टैक्सोनॉमी के लिए नए दृष्टिकोण". Annual Review of Microbiology. 17 (1): 329–372. doi:10.1146/annurev.mi.17.100163.001553. PMID 14147455.
- ↑ 4.0 4.1 Pace, N. R.; Sapp, J.; Goldenfeld, N. (17 January 2012). "Phylogeny and beyond: Scientific, historical, and conceptual significance of the first tree of life". Proceedings of the National Academy of Sciences. 109 (4): 1011–1018. Bibcode:2012PNAS..109.1011P. doi:10.1073/pnas.1109716109. PMC 3268332. PMID 22308526.
- ↑ Zuckerlandl, Emile; Pauling, Linus B. (1962). "आणविक रोग, विकास और आनुवंशिक विषमता". Horizons in Biochemistry: 189–225.
- ↑ Margoliash, E (Oct 1963). "साइटोक्रोम सी की प्राथमिक संरचना और विकास". Proc Natl Acad Sci U S A. 50 (4): 672–679. Bibcode:1963PNAS...50..672M. doi:10.1073/pnas.50.4.672. PMC 221244. PMID 14077496.
- ↑ Zuckerkandl, E; Pauling, LB (1965). विकासवादी विचलन और प्रोटीन में अभिसरण. pp. 96–166. doi:10.1016/B978-1-4832-2734-4.50017-6. ISBN 9781483227344.
{{cite book}}:|journal=ignored (help) - ↑ Eck, R. V.; Dayhoff, M. O. (15 April 1966). "आदिम अमीनो एसिड अनुक्रमों के जीवित अवशेषों के आधार पर फेरेडॉक्सिन की संरचना का विकास". Science. 152 (3720): 363–366. Bibcode:1966Sci...152..363E. doi:10.1126/science.152.3720.363. PMID 17775169. S2CID 23208558.
- ↑ Kimura, M (17 February 1968). "आणविक स्तर पर विकासवादी दर". Nature. 217 (5129): 624–626. Bibcode:1968Natur.217..624K. doi:10.1038/217624a0. PMID 5637732. S2CID 4161261.
- ↑ King, J. L.; Jukes, T. H. (16 May 1969). "गैर-डार्विनियन विकास". Science. 164 (3881): 788–798. Bibcode:1969Sci...164..788L. doi:10.1126/science.164.3881.788. PMID 5767777.
- ↑ Kimura, M; Ohta, T (1974). "आणविक विकास को नियंत्रित करने वाले कुछ सिद्धांतों पर". Proc Natl Acad Sci USA. 71 (7): 2848–2852. Bibcode:1974PNAS...71.2848K. doi:10.1073/pnas.71.7.2848. PMC 388569. PMID 4527913.
- ↑ Asthana, Saurabh; Roytberg, Mikhail; Stamatoyannopoulos, John; Sunyaev, Shamil (2007-12-28). Brudno, Michael (ed.). "न्यूक्लियोटाइड संकल्प पर अनुक्रम संरक्षण का विश्लेषण". PLOS Computational Biology (in English). 3 (12): e254. Bibcode:2007PLSCB...3..254A. doi:10.1371/journal.pcbi.0030254. ISSN 1553-7358. PMC 2230682. PMID 18166073.
- ↑ Cooper, G. M.; Brown, C. D. (2008-02-01). "अनुक्रम संरक्षण और आणविक कार्य के बीच संबंध को योग्य बनाना". Genome Research (in English). 18 (2): 201–205. doi:10.1101/gr.7205808. ISSN 1088-9051. PMID 18245453.
- ↑ Gilson, Amy I.; Marshall-Christensen, Ahmee; Choi, Jeong-Mo; Shakhnovich, Eugene I. (2017). "प्रोटीन संरचना विकास की गतिशीलता में विकासवादी चयन की भूमिका". Biophysical Journal (in English). 112 (7): 1350–1365. arXiv:1606.05802. Bibcode:2017BpJ...112.1350G. doi:10.1016/j.bpj.2017.02.029. PMC 5390048. PMID 28402878.
- ↑ Hunt, Ryan C.; Simhadri, Vijaya L.; Iandoli, Matthew; Sauna, Zuben E.; Kimchi-Sarfaty, Chava (2014). "पर्यायवाची उत्परिवर्तन को उजागर करना". Trends in Genetics (in English). 30 (7): 308–321. doi:10.1016/j.tig.2014.04.006. PMID 24954581.
- ↑ Zhang, Jianzhi (2000). "स्तनधारी परमाणु जीन में रूढ़िवादी और कट्टरपंथी गैर-पर्यायवाची न्यूक्लियोटाइड प्रतिस्थापन की दरें". Journal of Molecular Evolution (in English). 50 (1): 56–68. Bibcode:2000JMolE..50...56Z. doi:10.1007/s002399910007. ISSN 0022-2844. PMID 10654260. S2CID 15248867.
- ↑ Sousounis, Konstantinos; Haney, Carl E; Cao, Jin; Sunchu, Bharath; Tsonis, Panagiotis A (2012). "गैर-समरूप या असंबंधित प्रोटीन में त्रि-आयामी संरचना का संरक्षण". Human Genomics (in English). 6 (1): 10. doi:10.1186/1479-7364-6-10. ISSN 1479-7364. PMC 3500211. PMID 23244440.
- ↑ Kairys, Visvaldas; Fernandes, Miguel X. (2007). "SitCon: Binding site residue conservation visualization and protein sequence-to-function tool". International Journal of Quantum Chemistry. 107 (11): 2100–2110. Bibcode:2007IJQC..107.2100K. doi:10.1002/qua.21396. hdl:10400.13/5004. ISSN 0020-7608.
- ↑ Chamary, JV; Hurst, Laurence D (2005). "स्तनधारियों में mRNA माध्यमिक संरचना की स्थिरता को प्रभावित करने वाले पर्यायवाची उत्परिवर्तन पर चयन के लिए साक्ष्य". Genome Biology. 6 (9): R75. doi:10.1186/gb-2005-6-9-r75. PMC 1242210. PMID 16168082.
- ↑ Wadler, C. S.; Vanderpool, C. K. (27 November 2007). "A dual function for a bacterial small RNA: SgrS performs base pairing-dependent regulation and encodes a functional polypeptide". Proceedings of the National Academy of Sciences. 104 (51): 20454–20459. Bibcode:2007PNAS..10420454W. doi:10.1073/pnas.0708102104. PMC 2154452. PMID 18042713.
- ↑ Johnsson, Per; Lipovich, Leonard; Grandér, Dan; Morris, Kevin V. (March 2014). "Evolutionary conservation of long non-coding RNAs; sequence, structure, function". Biochimica et Biophysica Acta (BBA) - General Subjects. 1840 (3): 1063–1071. doi:10.1016/j.bbagen.2013.10.035. PMC 3909678. PMID 24184936.
- ↑ Freyhult, E. K.; Bollback, J. P.; Gardner, P. P. (6 December 2006). "Exploring genomic dark matter: A critical assessment of the performance of homology search methods on noncoding RNA". Genome Research. 17 (1): 117–125. doi:10.1101/gr.5890907. PMC 1716261. PMID 17151342.
- ↑ Margulies, E. H. (2003-12-01). "बहु-प्रजाति संरक्षित अनुक्रमों की पहचान और लक्षण वर्णन". Genome Research (in English). 13 (12): 2507–2518. doi:10.1101/gr.1602203. ISSN 1088-9051. PMC 403793. PMID 14656959.
- ↑ Edwards, John R.; Ruparel, Hameer; Ju, Jingyue (2005). "मास-स्पेक्ट्रोमेट्री डीएनए अनुक्रमण". Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis (in English). 573 (1–2): 3–12. doi:10.1016/j.mrfmmm.2004.07.021. PMID 15829234.
- ↑ Drost, Hajk-Georg; Gabel, Alexander; Grosse, Ivo; Quint, Marcel (2015-05-01). "पशु और पौधों के भ्रूणजनन में फाइलोट्रान्सक्रिपटोमिक ऑवरग्लास पैटर्न के सक्रिय रखरखाव के लिए साक्ष्य". Molecular Biology and Evolution (in English). 32 (5): 1221–1231. doi:10.1093/molbev/msv012. ISSN 0737-4038. PMC 4408408. PMID 25631928.
- ↑ Nawrocki, E. P.; Eddy, S. R. (4 September 2013). "Infernal 1.1: 100-fold faster RNA homology searches". Bioinformatics. 29 (22): 2933–2935. doi:10.1093/bioinformatics/btt509. PMC 3810854. PMID 24008419.
- ↑ Eddy, SR; Durbin, R (11 June 1994). "सहप्रसरण मॉडल का उपयोग करके आरएनए अनुक्रम विश्लेषण।". Nucleic Acids Research. 22 (11): 2079–88. doi:10.1093/nar/22.11.2079. PMC 308124. PMID 8029015.
- ↑ Trivedi, Rakesh; Nagarajaram, Hampapathalu Adimurthy (2020). "Substitution scoring matrices for proteins ‐ An overview". Protein Science (in English). 29 (11): 2150–2163. doi:10.1002/pro.3954. ISSN 0961-8368. PMC 7586916. PMID 32954566.
- ↑ 29.0 29.1 "वेबलॉग". UC Berkeley. Retrieved 30 December 2017.
- ↑ "क्लस्टल एफएक्यू #प्रतीक". Clustal. Archived from the original on 24 October 2016. Retrieved 8 December 2014.
- ↑ "ईसीआर ब्राउज़र". ईसीआर ब्राउज़र. Retrieved 9 January 2018.
- ↑ Earl, Dent; Nguyen, Ngan; Hickey, Glenn; Harris, Robert S.; Fitzgerald, Stephen; Beal, Kathryn; Seledtsov, Igor; Molodtsov, Vladimir; Raney, Brian J.; Clawson, Hiram; Kim, Jaebum; Kemena, Carsten; Chang, Jia-Ming; Erb, Ionas; Poliakov, Alexander; Hou, Minmei; Herrero, Javier; Kent, William James; Solovyev, Victor; Darling, Aaron E.; Ma, Jian; Notredame, Cedric; Brudno, Michael; Dubchak, Inna; Haussler, David; Paten, Benedict (December 2014). "Alignathon: a competitive assessment of whole-genome alignment methods". Genome Research. 24 (12): 2077–2089. doi:10.1101/gr.174920.114. PMC 4248324. PMID 25273068.
- ↑ Rouli, L.; Merhej, V.; Fournier, P.-E.; Raoult, D. (September 2015). "रोगजनक बैक्टीरिया के विश्लेषण के लिए एक नए उपकरण के रूप में बैक्टीरियल पैन्गेनोम". New Microbes and New Infections. 7: 72–85. doi:10.1016/j.nmni.2015.06.005. PMC 4552756. PMID 26442149.
- ↑ Méric, Guillaume; Yahara, Koji; Mageiros, Leonardos; Pascoe, Ben; Maiden, Martin C. J.; Jolley, Keith A.; Sheppard, Samuel K.; Bereswill, Stefan (27 March 2014). "A Reference Pan-Genome Approach to Comparative Bacterial Genomics: Identification of Novel Epidemiological Markers in Pathogenic Campylobacter". PLOS ONE. 9 (3): e92798. Bibcode:2014PLoSO...992798M. doi:10.1371/journal.pone.0092798. PMC 3968026. PMID 24676150.
- ↑ Cooper, G. M. (17 June 2005). "स्तनधारी जीनोमिक अनुक्रम में बाधा का वितरण और तीव्रता". Genome Research. 15 (7): 901–913. doi:10.1101/gr.3577405. PMC 1172034. PMID 15965027.
- ↑ "Sidow Lab - GERP".
- ↑ Nawar Malhis; Steven J. M. Jones; Jörg Gsponer (2019). "विकासवादी संरक्षण के लिए बेहतर उपाय जो टैक्सोनॉमी दूरियों का फायदा उठाते हैं". Nature Communications. 10 (1): 1556. Bibcode:2019NatCo..10.1556M. doi:10.1038/s41467-019-09583-2. PMC 6450959. PMID 30952844.
- ↑ Nawar Malhis; Matthew Jacobson; Steven J. M. Jones; Jörg Gsponer (2020). "LIST-S2: Taxonomy Based Sorting of Deleterious Missense Mutations Across Species". Nucleic Acids Research. 48 (W1): W154–W161. doi:10.1093/nar/gkaa288. PMC 7319545. PMID 32352516.
- ↑ Chang KT, Guo J, di Ronza A, Sardiello M (January 2018). "Aminode: Identification of Evolutionary Constraints in the Human Proteome". Sci. Rep. 8 (1): 1357. Bibcode:2018NatSR...8.1357C. doi:10.1038/s41598-018-19744-w. PMC 5778061. PMID 29358731.
- ↑ Pollard, K. S.; Hubisz, M. J.; Rosenbloom, K. R.; Siepel, A. (26 October 2009). "स्तनधारी फाइलोजेनियों पर गैर-तटस्थ प्रतिस्थापन दरों का पता लगाना". Genome Research. 20 (1): 110–121. doi:10.1101/gr.097857.109. PMC 2798823. PMID 19858363.
- ↑ "PHAST: Home".
- ↑ Fan, Xiaodan; Zhu, Jun; Schadt, Eric E; Liu, Jun S (2007). "क्रमिक रूप से संरक्षित तत्व का पता लगाने के लिए फाइलो-एचएमएम की सांख्यिकीय शक्ति". BMC Bioinformatics. 8 (1): 374. doi:10.1186/1471-2105-8-374. PMC 2194792. PMID 17919331.
- ↑ Bejerano, G. (28 May 2004). "मानव जीनोम में अतिसंरक्षित तत्व". Science. 304 (5675): 1321–1325. Bibcode:2004Sci...304.1321B. CiteSeerX 10.1.1.380.9305. doi:10.1126/science.1098119. PMID 15131266. S2CID 2790337.
- ↑ Siepel, A. (1 August 2005). "कशेरुकी, कीट, कृमि और खमीर जीनोम में क्रमिक रूप से संरक्षित तत्व". Genome Research. 15 (8): 1034–1050. doi:10.1101/gr.3715005. PMC 1182216. PMID 16024819.
- ↑ Harmston, N.; Baresic, A.; Lenhard, B. (11 November 2013). "अत्यधिक गैर-कोडिंग संरक्षण का रहस्य". Philosophical Transactions of the Royal Society B: Biological Sciences. 368 (1632): 20130021. doi:10.1098/rstb.2013.0021. PMC 3826495. PMID 24218634.
- ↑ Faircloth, B. C.; McCormack, J. E.; Crawford, N. G.; Harvey, M. G.; Brumfield, R. T.; Glenn, T. C. (9 January 2012). "अल्ट्राकंसर्व्ड एलिमेंट्स हजारों जेनेटिक मार्करों को कई इवोल्यूशनरी टाइमस्केल्स में फैलाते हैं". Systematic Biology. 61 (5): 717–726. doi:10.1093/sysbio/sys004. PMID 22232343.
- ↑ Faircloth, Brant C.; Branstetter, Michael G.; White, Noor D.; Brady, Seán G. (May 2015). "आर्थ्रोपोड्स से अतिसंरक्षित तत्वों का लक्ष्य संवर्धन हाइमनोप्टेरा के बीच संबंधों पर एक जीनोमिक परिप्रेक्ष्य प्रदान करता है". Molecular Ecology Resources. 15 (3): 489–501. doi:10.1111/1755-0998.12328. PMC 4407909. PMID 25207863.
- ↑ Reneker, J.; Lyons, E.; Conant, G. C.; Pires, J. C.; Freeling, M.; Shyu, C.-R.; Korkin, D. (10 April 2012). "पौधे और पशु जीनोम में लंबे समान बहु-प्रजाति तत्व". Proceedings of the National Academy of Sciences. 109 (19): E1183–E1191. doi:10.1073/pnas.1121356109. PMC 3358895. PMID 22496592.
- ↑ Isenbarger, Thomas A.; Carr, Christopher E.; Johnson, Sarah Stewart; Finney, Michael; Church, George M.; Gilbert, Walter; Zuber, Maria T.; Ruvkun, Gary (14 October 2008). "पृथ्वी और अन्य ग्रहों पर जीवन का पता लगाने के लिए सर्वाधिक संरक्षित जीनोम खंड". Origins of Life and Evolution of Biospheres. 38 (6): 517–533. Bibcode:2008OLEB...38..517I. doi:10.1007/s11084-008-9148-z. PMID 18853276. S2CID 15707806.
- ↑ Harris, J. K. (12 February 2003). "यूनिवर्सल पूर्वज का जेनेटिक कोर". Genome Research. 13 (3): 407–412. doi:10.1101/gr.652803. PMC 430263. PMID 12618371.
- ↑ Ban, Nenad; Beckmann, Roland; Cate, Jamie HD; Dinman, Jonathan D; Dragon, François; Ellis, Steven R; Lafontaine, Denis LJ; Lindahl, Lasse; Liljas, Anders; Lipton, Jeffrey M; McAlear, Michael A; Moore, Peter B; Noller, Harry F; Ortega, Joaquin; Panse, Vikram Govind; Ramakrishnan, V; Spahn, Christian MT; Steitz, Thomas A; Tchorzewski, Marek; Tollervey, David; Warren, Alan J; Williamson, James R; Wilson, Daniel; Yonath, Ada; Yusupov, Marat (February 2014). "राइबोसोमल प्रोटीन के नामकरण के लिए एक नई प्रणाली". Current Opinion in Structural Biology. 24: 165–169. doi:10.1016/j.sbi.2014.01.002. PMC 4358319. PMID 24524803.
- ↑ Gadagkar, Sudhindra R.; Rosenberg, Michael S.; Kumar, Sudhir (15 January 2005). "Inferring species phylogenies from multiple genes: Concatenated sequence tree versus consensus gene tree". Journal of Experimental Zoology Part B: Molecular and Developmental Evolution. 304B (1): 64–74. doi:10.1002/jez.b.21026. PMID 15593277.
- ↑ Ludwig, W; Schleifer, KH (October 1994). "Bacterial phylogeny based on 16S and 23S rRNA sequence analysis". FEMS Microbiology Reviews. 15 (2–3): 155–73. doi:10.1111/j.1574-6976.1994.tb00132.x. PMID 7524576.
- ↑ Hug, Laura A.; Baker, Brett J.; Anantharaman, Karthik; Brown, Christopher T.; Probst, Alexander J.; Castelle, Cindy J.; Butterfield, Cristina N.; Hernsdorf, Alex W.; Amano, Yuki; Ise, Kotaro; Suzuki, Yohey; Dudek, Natasha; Relman, David A.; Finstad, Kari M.; Amundson, Ronald; Thomas, Brian C.; Banfield, Jillian F. (11 April 2016). "जीवन के वृक्ष का एक नया दृश्य". Nature Microbiology. 1 (5): 16048. doi:10.1038/nmicrobiol.2016.48. PMID 27572647.
- ↑ Zhang, Liqing; Li, Wen-Hsiung (February 2004). "स्तनधारी हाउसकीपिंग जीन ऊतक-विशिष्ट जीन की तुलना में अधिक धीरे-धीरे विकसित होते हैं". Molecular Biology and Evolution. 21 (2): 236–239. doi:10.1093/molbev/msh010. PMID 14595094.
- ↑ Clermont, O.; Bonacorsi, S.; Bingen, E. (1 October 2000). "एस्चेरिचिया कोलाई फाइलोजेनेटिक ग्रुप का रैपिड एंड सिंपल निर्धारण". Applied and Environmental Microbiology. 66 (10): 4555–4558. Bibcode:2000ApEnM..66.4555C. doi:10.1128/AEM.66.10.4555-4558.2000. PMC 92342. PMID 11010916.
- ↑ Kullberg, Morgan; Nilsson, Maria A.; Arnason, Ulfur; Harley, Eric H.; Janke, Axel (August 2006). "यूथेरियन संबंधों के फाइलोजेनेटिक विश्लेषण के लिए हाउसकीपिंग जीन". Molecular Biology and Evolution. 23 (8): 1493–1503. doi:10.1093/molbev/msl027. PMID 16751257.
- ↑ Schoch, C. L.; Seifert, K. A.; Huhndorf, S.; Robert, V.; Spouge, J. L.; Levesque, C. A.; Chen, W.; Bolchacova, E.; Voigt, K.; Crous, P. W.; Miller, A. N.; Wingfield, M. J.; Aime, M. C.; An, K.-D.; Bai, F.-Y.; Barreto, R. W.; Begerow, D.; Bergeron, M.-J.; Blackwell, M.; Boekhout, T.; Bogale, M.; Boonyuen, N.; Burgaz, A. R.; Buyck, B.; Cai, L.; Cai, Q.; Cardinali, G.; Chaverri, P.; Coppins, B. J.; Crespo, A.; Cubas, P.; Cummings, C.; Damm, U.; de Beer, Z. W.; de Hoog, G. S.; Del-Prado, R.; Dentinger, B.; Dieguez-Uribeondo, J.; Divakar, P. K.; Douglas, B.; Duenas, M.; Duong, T. A.; Eberhardt, U.; Edwards, J. E.; Elshahed, M. S.; Fliegerova, K.; Furtado, M.; Garcia, M. A.; Ge, Z.-W.; Griffith, G. W.; Griffiths, K.; Groenewald, J. Z.; Groenewald, M.; Grube, M.; Gryzenhout, M.; Guo, L.-D.; Hagen, F.; Hambleton, S.; Hamelin, R. C.; Hansen, K.; Harrold, P.; Heller, G.; Herrera, C.; Hirayama, K.; Hirooka, Y.; Ho, H.-M.; Hoffmann, K.; Hofstetter, V.; Hognabba, F.; Hollingsworth, P. M.; Hong, S.-B.; Hosaka, K.; Houbraken, J.; Hughes, K.; Huhtinen, S.; Hyde, K. D.; James, T.; Johnson, E. M.; Johnson, J. E.; Johnston, P. R.; Jones, E. B. G.; Kelly, L. J.; Kirk, P. M.; Knapp, D. G.; Koljalg, U.; Kovacs, G. M.; Kurtzman, C. P.; Landvik, S.; Leavitt, S. D.; Liggenstoffer, A. S.; Liimatainen, K.; Lombard, L.; Luangsa-ard, J. J.; Lumbsch, H. T.; Maganti, H.; Maharachchikumbura, S. S. N.; Martin, M. P.; May, T. W.; McTaggart, A. R.; Methven, A. S.; Meyer, W.; Moncalvo, J.-M.; Mongkolsamrit, S.; Nagy, L. G.; Nilsson, R. H.; Niskanen, T.; Nyilasi, I.; Okada, G.; Okane, I.; Olariaga, I.; Otte, J.; Papp, T.; Park, D.; Petkovits, T.; Pino-Bodas, R.; Quaedvlieg, W.; Raja, H. A.; Redecker, D.; Rintoul, T. L.; Ruibal, C.; Sarmiento-Ramirez, J. M.; Schmitt, I.; Schussler, A.; Shearer, C.; Sotome, K.; Stefani, F. O. P.; Stenroos, S.; Stielow, B.; Stockinger, H.; Suetrong, S.; Suh, S.-O.; Sung, G.-H.; Suzuki, M.; Tanaka, K.; Tedersoo, L.; Telleria, M. T.; Tretter, E.; Untereiner, W. A.; Urbina, H.; Vagvolgyi, C.; Vialle, A.; Vu, T. D.; Walther, G.; Wang, Q.-M.; Wang, Y.; Weir, B. S.; Weiss, M.; White, M. M.; Xu, J.; Yahr, R.; Yang, Z. L.; Yurkov, A.; Zamora, J.-C.; Zhang, N.; Zhuang, W.-Y.; Schindel, D. (27 March 2012). "कवक के लिए एक सार्वभौमिक डीएनए बारकोड मार्कर के रूप में परमाणु राइबोसोमल आंतरिक लिखित स्पेसर (ITS) क्षेत्र". Proceedings of the National Academy of Sciences. 109 (16): 6241–6246. doi:10.1073/pnas.1117018109. PMC 3341068. PMID 22454494.
- ↑ Man, S. M.; Kaakoush, N. O.; Octavia, S.; Mitchell, H. (26 March 2010). "आंतरिक लिखित स्पेसर क्षेत्र, कैंपिलोबैक्टर जीनस के भीतर प्रजातियों के भेदभाव और व्यवस्थित संबंधों के चित्रण में उपयोग के लिए एक नया उपकरण". Applied and Environmental Microbiology. 76 (10): 3071–3081. Bibcode:2010ApEnM..76.3071M. doi:10.1128/AEM.02551-09. PMC 2869123. PMID 20348308.
- ↑ Ranjard, L.; Poly, F.; Lata, J.-C.; Mougel, C.; Thioulouse, J.; Nazaret, S. (1 October 2001). "Characterization of Bacterial and Fungal Soil Communities by Automated Ribosomal Intergenic Spacer Analysis Fingerprints: Biological and Methodological Variability". Applied and Environmental Microbiology. 67 (10): 4479–4487. Bibcode:2001ApEnM..67.4479R. doi:10.1128/AEM.67.10.4479-4487.2001. PMC 93193. PMID 11571146.
- ↑ Bidet, Philippe; Barbut, Frédéric; Lalande, Valérie; Burghoffer, Béatrice; Petit, Jean-Claude (June 1999). "राइबोसोमल आरएनए जीन अनुक्रमण के आधार पर एक नई पीसीआर-राइबोटाइपिंग पद्धति का विकास". FEMS Microbiology Letters. 175 (2): 261–266. doi:10.1111/j.1574-6968.1999.tb13629.x. PMID 10386377.
- ↑ Ala, Ugo; Piro, Rosario Michael; Grassi, Elena; Damasco, Christian; Silengo, Lorenzo; Oti, Martin; Provero, Paolo; Di Cunto, Ferdinando; Tucker-Kellogg, Greg (28 March 2008). "मानव-माउस संरक्षित सह-अभिव्यक्ति विश्लेषण द्वारा मानव रोग जीन की भविष्यवाणी". PLOS Computational Biology. 4 (3): e1000043. Bibcode:2008PLSCB...4E0043A. doi:10.1371/journal.pcbi.1000043. PMC 2268251. PMID 18369433.
- ↑ Pandey, U. B.; Nichols, C. D. (17 March 2011). "ड्रोसोफिला मेलानोगास्टर में मानव रोग मॉडल और चिकित्सीय ड्रग डिस्कवरी में मक्खी की भूमिका". Pharmacological Reviews. 63 (2): 411–436. doi:10.1124/pr.110.003293. PMC 3082451. PMID 21415126.
- ↑ Huang, Hui; Winter, Eitan E; Wang, Huajun; Weinstock, Keith G; Xing, Heming; Goodstadt, Leo; Stenson, Peter D; Cooper, David N; Smith, Douglas; Albà, M Mar; Ponting, Chris P; Fechtel, Kim (2004). "चूहे और चूहे के जीनोम में मानव रोग जीन ऑर्थोलॉग का विकासवादी संरक्षण और चयन". Genome Biology. 5 (7): R47. doi:10.1186/gb-2004-5-7-r47. PMC 463309. PMID 15239832.
- ↑ Ge, Dongliang; Fellay, Jacques; Thompson, Alexander J.; Simon, Jason S.; Shianna, Kevin V.; Urban, Thomas J.; Heinzen, Erin L.; Qiu, Ping; Bertelsen, Arthur H.; Muir, Andrew J.; Sulkowski, Mark; McHutchison, John G.; Goldstein, David B. (16 August 2009). "Genetic variation in IL28B predicts hepatitis C treatment-induced viral clearance". Nature. 461 (7262): 399–401. Bibcode:2009Natur.461..399G. doi:10.1038/nature08309. PMID 19684573. S2CID 1707096.
- ↑ Bertram, L. (2009). "अल्जाइमर रोग में जीनोम-वाइड एसोसिएशन अध्ययन". Human Molecular Genetics. 18 (R2): R137–R145. doi:10.1093/hmg/ddp406. PMC 2758713. PMID 19808789.
- ↑ Kellis, Manolis; Patterson, Nick; Endrizzi, Matthew; Birren, Bruce; Lander, Eric S. (15 May 2003). "जीन और नियामक तत्वों की पहचान करने के लिए खमीर प्रजातियों की अनुक्रमण और तुलना". Nature. 423 (6937): 241–254. Bibcode:2003Natur.423..241K. doi:10.1038/nature01644. PMID 12748633. S2CID 1530261.
- ↑ Marchler-Bauer, A.; Lu, S.; Anderson, J. B.; Chitsaz, F.; Derbyshire, M. K.; DeWeese-Scott, C.; Fong, J. H.; Geer, L. Y.; Geer, R. C.; Gonzales, N. R.; Gwadz, M.; Hurwitz, D. I.; Jackson, J. D.; Ke, Z.; Lanczycki, C. J.; Lu, F.; Marchler, G. H.; Mullokandov, M.; Omelchenko, M. V.; Robertson, C. L.; Song, J. S.; Thanki, N.; Yamashita, R. A.; Zhang, D.; Zhang, N.; Zheng, C.; Bryant, S. H. (24 November 2010). "CDD: a Conserved Domain Database for the functional annotation of proteins". Nucleic Acids Research. 39 (Database): D225–D229. doi:10.1093/nar/gkq1189. PMC 3013737. PMID 21109532.