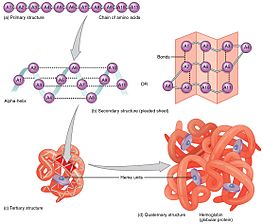
प्रोटीन वलन
प्रोटीन वलन एक ऐसी भौतिक प्रक्रिया है, जिसके द्वारा एक प्रोटीन श्रृंखला को उसके मूल त्रि-आयामी संरचना में अनुवादित (जीव विज्ञान) किया जाता है, सामान्य रूप से एक वलित संरचना जिसके द्वारा प्रोटीन जैविक रूप से क्रियाशील हो जाता है। तथा एक त्वरित और पुनरुत्पादनीय प्रक्रिया के माध्यम से, एक पॉलीपेप्टाइड एक यादृच्छिक कुण्डली से अपनी विशिष्ट त्रि-आयामी संरचना में परिवर्तित हो जाता है।[1] mRNA के एक अनुक्रम से अमीनो अम्ल की एक रैखिक श्रृंखला में अनुवादित होने के बाद प्रत्येक प्रोटीन से पहले एक सामने आया पॉलीपेप्टाइड या यादृच्छिक कुंडल के रूप में उपस्थित होता है। इस स्तर पर पॉलीपेप्टाइड में किसी भी स्थिर (लंबे समय तक चलने वाली) त्रि-आयामी संरचना (पहली आकृति के बाएं हाथ की ओर) का अभाव होता है। जैसा कि पॉलीपेप्टाइड श्रृंखला को राइबोसोम द्वारा संश्लेषित किया जा रहा है, रैखिक श्रृंखला इसकी त्रि-आयामी संरचना में परिवर्तित होना प्रारम्भ कर देती है।
पॉलीपेप्टाइड श्रृंखला के अनुवादन के दौरान भी कई प्रोटीनों का वलन प्रारम्भ हो जाता है। अमीनो अम्ल एक दूसरे के साथ एक अच्छी तरह से परिभाषित त्रि-आयामी संरचना वलित प्रोटीन (आकृति के दाहिने हाथ की ओर), जिसे मूल अवस्था के रूप में जाना जाता है, जिसका उत्पादन करने के लिए परस्पर प्रभाव करते हैं। परिणामी त्रि-आयामी संरचना अमीनो अम्ल अनुक्रम या प्राथमिक संरचना (एनफिन्सन सिद्धांत) द्वारा निर्धारित की जाती है।[2]
कार्य करने के लिए सही त्रि-आयामी संरचना आवश्यक होती है, हालांकि कार्यात्मक प्रोटीन के कुछ भाग प्रकट हो सकते हैं, [3] ताकि प्रोटीन गतिशीलता महत्वपूर्ण हो। प्राकृतिक संरचना में वलन में विफलता सामान्य रूप से निष्क्रिय प्रोटीन का उत्पादन करती है, लेकिन कुछ स्थितियों में मिसफॉल्ड प्रोटीन में संशोधित या विषाक्त कार्यक्षमता होती है। माना जाता है कि कई न्यूरोडीजेनेरेटिव और अन्य बीमारियां मिसफोल्डेड प्रोटीन द्वारा गठित अमाइलॉइड फाइब्रिल के संचय के परिणामस्वरूप होती हैं, जिनमें से संक्रामक किस्मों को प्रोटीन संक्रमण के रूप में जाना जाता है।[4] कई एलर्जी कुछ प्रोटीनों की गलत वलन के कारण होती हैं, क्योंकि उन्मुक्त प्रणाली कुछ प्रोटीन संरचनाओं के लिए एंटीबॉडी का उत्पादन नहीं करती है।[5]
प्रोटीन का विकृतीकरण (जैव रसायन) वलित से बिना वलित अवस्था में संक्रमण की एक प्रक्रिया होती है। यह खाना पकाने में, जलने में, प्रोटीनोपैथियों में और अन्य संदर्भों में होता है।
वलन प्रक्रिया का समयांतराल प्रेरित प्रोटीन के आधार पर प्रभावशाली तरीके से भिन्न होता है। जब कोशिका के बाहर अध्ययन किया जाता है, तो सबसे धीमी गति से वलन वाले प्रोटीन को मुख्य रूप से प्रोलीन समावयवन के कारण वलन में कई मिनट या घंटे लगते हैं, और प्रक्रिया पूरी होने से पहले, कई मध्यवर्ती अवस्थाओं जैसे चौकियों(नाका) से गुजरना पड़ता है।[6] दूसरी ओर, सौ अमीनो अम्ल तक की लंबाई वाले बहुत छोटे एकल-डोमेन प्रोटीन सामान्य रूप से एक ही चरण में वलित हो जाते हैं।[7] मिलीसेकेंड का समय पैमाना मानक है और सबसे तेज़ ज्ञात प्रोटीन वलन प्रतिक्रियाएं कुछ माइक्रोसेकंड के भीतर पूरी हो जाती हैं।[8] एक प्रोटीन का वलन कालक्रम उसके आकार, संपर्क क्रम और चक्रण टोपोलॉजी पर निर्भर करता है।[9]
1960 के दशक के उत्तरार्ध से कम्प्यूटेशनल बायोलॉजी विज्ञान के लिए प्रोटीन वलन प्रक्रिया को समझना और अनुकरण करना एक महत्वपूर्ण चुनौती रही है।
प्रोटीन वलन की प्रक्रिया
प्राथमिक संरचना
एक प्रोटीन की प्राथमिक संरचना, इसका रैखिक अमीनो-अम्ल अनुक्रम, इसकी मूल संरचना को निर्धारित करता है।[10] विशिष्ट अमीनो अम्ल अवशेष और पॉलीपेप्टाइड श्रृंखला में उनकी स्थिति निर्धारित करने वाले कारक हैं, जिनके लिए प्रोटीन के एक हिस्से साथ जुड़ते हैं और इसकी त्रि-आयामी संरचना को बनाते हैं। तथा अमीनो अम्ल की संरचना क्रम की तरह महत्वपूर्ण नहीं होते है।[11] हालांकि, वलन का आवश्यक तथ्य यह है कि प्रत्येक प्रोटीन के अमीनो अम्ल अनुक्रम में वह जानकारी होती है, जो उस स्थिति को प्राप्त करने के लिए मूल संरचना और मार्ग दोनों को निर्दिष्ट करती है। इसका अर्थ यह नहीं है कि लगभग समान अमीनो अम्ल अनुक्रम हमेशा समान रूप से वलित होते हैं। रेफरी>Alexander PA, He Y, Chen Y, Orban J, Bryan PN (July 2007). "88% अनुक्रम पहचान लेकिन विभिन्न संरचना और कार्य के साथ दो प्रोटीनों का डिज़ाइन और लक्षण वर्णन". Proceedings of the National Academy of Sciences of the United States of America. 104 (29): 11963–8. Bibcode:2007PNAS..10411963A. doi:10.1073/pnas.0700922104. PMC 1906725. PMID 17609385.</ref> अनुकूलता पर्यावरणीय कारकों के आधार पर भी भिन्न होती है। तथा जहां वे पाए जाते हैं, उसके आधार पर समान प्रोटीन अलग-अलग वलन मे होते हैं।
माध्यमिक संरचना

एक द्वितीयक संरचना का निर्माण वलन प्रक्रिया में पहला चरण होता है, जिसे एक प्रोटीन अपनी मूल संरचना ग्रहण करने के लिए लेता है। द्वितीयक संरचना की विशेषता वे संरचनाएँ होती हैं जिन्हें अल्फा हेलिक्स और बीटा शीट्स के रूप में जाना जाता है, जो तेजी से वलित होती हैं क्योंकि वे आंतरआण्विक बल, हाइड्रोजन बंध द्वारा स्थिर होती हैं, जैसा कि पहली बार लिनुस पॉलिंग द्वारा किया गया था। आंतरआण्विक हाइड्रोजन बंध का निर्माण प्रोटीन स्थिरता में एक और महत्वपूर्ण योगदान प्रदान करता है।[12] α-हेलिक्स रीढ़ की हड्डी के हाइड्रोजन बंधन द्वारा एक कुंडली का आकार बनाने के लिए बनते हैं। (दाईं ओर की आकृति देखें)।[11] β प्लीटेड शीट्स एक संरचना होती है, जो हाइड्रोजन बन्ध बनाने के लिए रीढ़ की हड्डी के साथ स्वय को झुकाती है (जैसा कि बाईं ओर की आकृति में दिखाया गया है)। हाइड्रोजन बंध पेप्टाइड बंधन के ऐमाइड हाइड्रोजन और कार्बोनिल ऑक्सीजन के बीच होते हैं। एंटी-पैरेलल β प्लीटेड शीट्स और समानांतर β प्लीटेड शीट्स उपस्थित हैं, जहां हाइड्रोजन बंध की स्थिरता एंटी-पैरलल β शीट्स में जटिल होती है क्योंकि यह समानांतर शीट्स द्वारा बनाए गए झुके हुए हाइड्रोजन बंध की तुलना में आदर्श 180 डिग्री के कोण के साथ हाइड्रोजन बंध होते हैं। [11]
तृतीयक संरचना
α-हेलिक्स और β-शीट्स सामान्य रूप से एम्फीपैथिक होते हैं, जिसका अर्थ है कि उनके पास एक हाइड्रोफिलिक और एक हाइड्रोफोबिक भाग होता है। यह क्षमता एक प्रोटीन की तृतीयक संरचना बनाने में मदद करती है जिसमें वलन होता है ताकि हाइड्रोफिलिक पक्ष प्रोटीन के आसपास के जलीय वातावरण का सामना कर रहे हों और हाइड्रोफोबिक पक्ष प्रोटीन के हाइड्रोफोबिक भीतरी भाग का सामना कर रहे हों।[13] द्वितीयक संरचना श्रेणीबद्ध रूप से तृतीयक संरचना निर्माण का मार्ग प्रशस्त करती है। एक बार जब प्रोटीन की तृतीयक संरचना हाइड्रोफोबिक से परस्पर क्रिया द्वारा बनती और स्थिर हो जाती है, तो दो सिस्टिइन अवशेषों के बीच बने डाइसल्फ़ाइड बंधन के रूप में सहसंयोजक बंधन भी हो सकते हैं। ये गैर-सहसंयोजक और सहसंयोजक संपर्क एक प्रोटीन की मूल संरचना में एक विशिष्ट स्थलीय अवस्था लेते हैं। प्रोटीन की तृतीयक संरचना में एकल पॉलीपेप्टाइड श्रृंखला सम्मिलित होती है। हालांकि, वलन पॉलीपेप्टाइड श्रृंखलाओं की अतिरिक्त अंतःक्रियाएं चतुर्धातुक संरचना निर्माण को जन्म देती हैं।[14]
चतुर्धातुक संरचना
तृतीयक संरचना कुछ प्रोटीनों में चतुर्धातुक संरचना के निर्माण के लिए मार्ग दे सकती है, जिसमें सामान्य रूप से पहले से वलित सबयूनिट्स का समन्वायोजन या उपसमन्वायोजन सम्मिलित होता है। दूसरे शब्दों में, बहु पॉलीपेप्टाइड शृंखलाएं परस्पर क्रिया करके एक पूर्णतया क्रियाशील चतुर्धातुक प्रोटीन का निर्माण कर सकती हैं।[11]
प्रोटीन वलन की प्रेरक शक्ति
वलन एक सहज प्रक्रिया है जो मुख्य रूप से हाइड्रोफोबिक इंटरैक्शन, इंट्रामोल्युलर हाइड्रोजन बॉन्ड के गठन, वैन डेर वाल्स बलों द्वारा निर्देशित होती है, और यह कंफॉर्मल एन्ट्रापी द्वारा विरोध किया जाता है।[15] वलन की प्रक्रिया अधिकांश सह-अनुवादिक रूप से प्रारम्भ होती है, जिससे प्रोटीन का N- टर्मिनस मुड़ना प्रारम्भ हो जाता है जबकि प्रोटीन का सी-टर्मिनल हिस्सा अभी भी राइबोसोम द्वारा संश्लेषित किया जा रहा है; हालांकि, जैवसंश्लेषण के दौरान या बाद में एक प्रोटीन अणु अनायास मुड़ सकता है।[16] जबकि इन मैक्रो मोलेक्यूल को स्व विधानसभा के रूप में माना जा सकता है, यह प्रक्रिया विलायक (पानी या लिपिड बिलेयर), लवण की एकाग्रता (रसायन विज्ञान), पीएच, तापमान, कॉफ़ेक्टर्स की संभावित उपस्थिति और आणविक चैपरोन (प्रोटीन) पर भी निर्भर करती है।[17]
सीमित झुकने वाले कोणों या संभव होने वाले अनुरूपणों द्वारा प्रोटीन की अपनी वलन क्षमताओं पर सीमाएं होंगी। प्रोटीन वलन के इन स्वीकार्य कोणों को एक द्वि-आयामी प्लॉट के साथ वर्णित किया गया है जिसे रामचंद्रन प्लॉट के रूप में जाना जाता है, जिसे स्वीकार्य रोटेशन के साई और फाई कोणों के साथ दर्शाया गया है।[18]
हाइड्रोफोबिक प्रभाव
एक सहज प्रतिक्रिया होने के लिए प्रोटीन वलन को सेल के भीतर थर्मोडायनामिक रूप से अनुकूल होना चाहिए। चूंकि यह ज्ञात है कि प्रोटीन वलन एक सहज प्रतिक्रिया है, तो इसे एक नकारात्मक गिब्स मुक्त ऊर्जा मूल्य मान लेना चाहिए। प्रोटीन वलन में गिब्स मुक्त ऊर्जा का सीधा संबंध एन्थैल्पी और एन्ट्रापी से है।[11] एक नकारात्मक डेल्टा G उत्पन्न होने के लिए और प्रोटीन वलन के लिए थर्मोडायनामिक रूप से अनुकूल बनने के लिए, या तो एन्थैल्पी, एंट्रॉपी, या दोनों शर्तें अनुकूल होनी चाहिए।
पानी के संपर्क में आने वाली हाइड्रोफोबिक साइड-चेन की संख्या को कम करना वलन प्रक्रिया के पीछे एक महत्वपूर्ण प्रेरक शक्ति है।[19] हाइड्रोफोबिक प्रभाव वह परिघटना है जिसमें प्रोटीन की हाइड्रोफोबिक श्रृंखलाएं प्रोटीन के कोर (हाइड्रोफिलिक वातावरण से दूर) में ढह जाती हैं।[11] एक जलीय वातावरण में, पानी के अणु हाइड्रोफोबिक क्षेत्रों या प्रोटीन की साइड चेन के चारों ओर एकत्रित होते हैं, जिससे पानी के अणुओं के पानी के गोले बनते हैं।[20]
एक हाइड्रोफोबिक क्षेत्र के आसपास पानी के अणुओं का क्रम एक प्रणाली में क्रम बढ़ाता है और इसलिए एंट्रॉपी (सिस्टम में कम एन्ट्रापी) में नकारात्मक परिवर्तन का योगदान देता है। पानी के अणु इन पानी के पिंजरों में तय होते हैं जो हाइड्रोफोबिक पतन, या हाइड्रोफोबिक समूहों के अंदरूनी वलन को चलाते हैं। हाइड्रोफोबिक कोलैप्स पानी के पिंजरों को तोड़कर प्रणाली में एन्ट्रापी को वापस लाता है जो पानी के अणुओं को मुक्त करता है।[11] ग्लोबुलर फोल्डेड प्रोटीन के कोर के भीतर परस्पर क्रिया करने वाले हाइड्रोफोबिक समूहों की भीड़, वलन के बाद प्रोटीन स्थिरता के लिए महत्वपूर्ण मात्रा में योगदान करती है, क्योंकि बड़े पैमाने पर वैन डेर वाल्स बल (विशेष रूप से लंडन फैलाव बल) जमा होते हैं।[11] उष्मप्रवैगिकी में हाइड्रोफोबिक प्रभाव एक प्रेरक शक्ति के रूप में तभी मौजूद होता है जब एक बड़े हाइड्रोफोबिक क्षेत्र वाले एम्फीफिलिक अणु के साथ एक जलीय माध्यम की उपस्थिति होती है।[21] हाइड्रोजन बन्ध की ताकत उनके पर्यावरण पर निर्भर करती है; इस प्रकार, हाइड्रोफोबिक कोर में लिपटे एच-बन्ध मूल राज्य की स्थिरता के लिए जलीय पर्यावरण के संपर्क में आने वाले एच-बॉन्ड से अधिक योगदान करते हैं।[22]
गोलाकार वलनों वाले प्रोटीनों में, हाइड्रोफोबिक अमीनो अम्ल बेतरतीब ढंग से वितरित या एक साथ गुच्छित होने के बजाय, प्राथमिक अनुक्रम में बीच-बीच में बिखर जाते हैं।[23][24] हालांकि, प्रोटीन जो हाल ही में नए सिरे से पैदा हुए हैं, जो आंतरिक रूप से अव्यवस्थित प्रोटीन होते हैं,[25][26] प्राथमिक अनुक्रम के साथ हाइड्रोफोबिक अमीनो अम्ल क्लस्टरिंग के विपरीत पैटर्न दिखाते हैं।[27]
चैपरोन
चैपेरोन (प्रोटीन) प्रोटीन का एक वर्ग है जो विवो में अन्य प्रोटीनों के सही वलन में सहायता करता है। चैपरोन सभी सेलुलर डिब्बों में मौजूद होते हैं और प्रोटीन के मूल त्रि-आयामी संचलन की अनुमति देने के लिए पॉलीपेप्टाइड श्रृंखला के साथ बातचीत करते हैं; हालाँकि, संरक्षक स्वयं उस प्रोटीन की अंतिम संरचना में सम्मिलित नहीं होते हैं जिसमें वे सहायता कर रहे हैं।[28]जब नवजात पॉलीपेप्टाइड को राइबोसोम द्वारा संश्लेषित किया जा रहा हो तब भी चैपरोन वलन में सहायता कर सकते हैं।[29]आणविक संरक्षिकाएं अपने वलन मार्ग में एक प्रोटीन की अन्यथा अस्थिर संरचना को स्थिर करने के लिए बाध्यकारी द्वारा संचालित होती हैं, लेकिन संरक्षिकाओं में प्रोटीन की सही मूल संरचना को जानने के लिए आवश्यक जानकारी नहीं होती है जो वे सहायता कर रहे हैं; बल्कि, गलत वलन कन्फर्मेशन को रोककर चैपरोन काम करते हैं।Cite error: Closing </ref> missing for <ref> tag
एक पूरी तरह से विकृत प्रोटीन में तृतीयक और द्वितीयक संरचना दोनों का अभाव होता है, और एक तथाकथित यादृच्छिक कुंडल के रूप में मौजूद होता है। कुछ शर्तों के वलनत कुछ प्रोटीन रिफोल्ड हो सकते हैं; हालाँकि, कई स्थितियों में, विकृतीकरण अपरिवर्तनीय है।[30] कोशिकाएं कभी-कभी हीट शॉक प्रोटीन (एक प्रकार का चैपरोन) के रूप में जाने वाले एंजाइम के साथ गर्मी के विकृतीकरण प्रभाव के खिलाफ अपने प्रोटीन की रक्षा करती हैं, जो अन्य प्रोटीनों को वलन और शेष मुड़ने में सहायता करती हैं। जीवाणुओं से लेकर मनुष्यों तक, जांच की गई सभी प्रजातियों में हीट शॉक प्रोटीन पाए गए हैं, जो यह सुझाव देते हैं कि वे बहुत जल्दी विकसित हुए और एक महत्वपूर्ण कार्य करते हैं। कुछ प्रोटीन कोशिकाओं में बिल्कुल भी मुड़ते नहीं हैं सिवाय चैपरोन की सहायता से जो या तो अलग-अलग प्रोटीन को अलग कर देते हैं ताकि उनका वलन अन्य प्रोटीन के साथ बातचीत से बाधित न हो या मिसफोल्डेड प्रोटीन को प्रकट करने में मदद करे, जिससे वे सही मूल संरचना में फिर से जुड़ सकें।[31] और यहां तक कि स्थान की सीमा (अर्थात् कारावास), जो प्रोटीन की वलन पर बड़ा प्रभाव डाल सकता है। रेफरी>Ellis RJ (July 2006). "मॉलिक्यूलर चैपरोन: फोल्डिंग के अलावा असिस्टिंग असेंबली". Trends in Biochemical Sciences. 31 (7): 395–401. doi:10.1016/j.tibs.2006.05.001. PMID 16716593.</ रेफ> विलेय की उच्च सांद्रता, चरम पीएच, यांत्रिक बल, और रासायनिक विकृतीकरण की उपस्थिति प्रोटीन विकृतीकरण में भी योगदान दे सकती है। इन व्यक्तिगत कारकों को तनाव के रूप में एक साथ वर्गीकृत किया गया है। कोशिकीय तनाव के समय चैपरोनों की बढ़ती सांद्रता में मौजूद होने को दिखाया गया है और उभरते हुए प्रोटीनों के साथ-साथ विकृत या गलत तरीके से वलन में मदद करता है।[28]
कुछ स्थितियों में प्रोटीन अपने जैवरासायनिक रूप से कार्यात्मक रूपों में नहीं मुड़ेंगे। उस सीमा से ऊपर या नीचे तापमान जिसमें कोशिकाएं रहती हैं, थर्मोस्टेबिलिटी प्रोटीन को प्रकट या विकृत करने का कारण बनेगी (यही कारण है कि उबालने से अंडे का सफेद # विकृतीकरण अपारदर्शी हो जाता है)। हालांकि, प्रोटीन थर्मल स्थिरता स्थिर से बहुत दूर है; उदाहरण के लिए, hyperthermophiles पाए गए हैं जो 122 °C तक के उच्च तापमान पर बढ़ते हैं,[32] निश्चित रूप से यह आवश्यक है कि महत्वपूर्ण प्रोटीन और प्रोटीन असेंबली का उनका पूरा पूरक उस तापमान या उससे ऊपर स्थिर हो।
जीवाणु एस्चेरिचिया कोलाई | ई। कोली एस्चेरिचिया वायरस T4 4 के लिए मेजबान है, और फेज एन्कोडेड जीपी31 प्रोटीन (P17313) ई. कोलाई चैपरोन (प्रोटीन) ग्रॉस के लिए संरचनात्मक और कार्यात्मक रूप से समरूप प्रतीत होता है और संक्रमण के दौरान बैक्टीरियोफेज टी 4 वाइरस कणों की असेंबली में इसके लिए स्थानापन्न करने में सक्षम है।[33] GroES की तरह, gp31 ग्रेल चैपरोनिन के साथ एक स्थिर कॉम्प्लेक्स बनाता है जो बैक्टीरियोफेज T4 प्रमुख कैप्सिड प्रोटीन gp23 के विवो में वलन और असेंबली के लिए बिल्कुल जरूरी है।[33]
फोल्ड स्विचिंग
कुछ प्रोटीनों में कई मूल संरचनाएं होती हैं, और कुछ बाहरी कारकों के आधार पर उनकी वलन बदल जाती है। उदाहरण के लिए, काईबी प्रोटीन काईबी #सर्कैडियन आउटपुट और काईबी फोल्ड स्विचिंग, साइनोबैक्टीरिया के लिए घड़ी के रूप में कार्य करता है। यह अनुमान लगाया गया है कि लगभग 0.5-4% पीडीबी (प्रोटीन डाटा बैंक) प्रोटीन फोल्ड हो जाते हैं।[34]
प्रोटीन मिसफॉल्डिंग और न्यूरोडीजेनेरेटिव रोग
एक प्रोटीन को प्रोटीन मिसफॉल्डिंग माना जाता है यदि वह अपनी सामान्य मूल अवस्था को प्राप्त नहीं कर पाता है। यह अमीनो अम्ल अनुक्रम में उत्परिवर्तन या बाहरी कारकों द्वारा सामान्य वलन प्रक्रिया में व्यवधान के कारण हो सकता है।[35]मिसफोल्डेड प्रोटीन में सामान्य रूप से बीटा शीट | β-शीट होती हैं जो एक सुपरमॉलेक्यूलर व्यवस्था में व्यवस्थित होती हैं जिसे क्रॉस-β संरचना के रूप में जाना जाता है। ये β-शीट-रिच असेंबली बहुत स्थिर, बहुत अघुलनशील और सामान्य रूप से प्रोटियोलिसिस के प्रतिरोधी हैं।Cite error: Closing </ref> missing for <ref> tag साथ ही हंटिंगटन और पार्किंसंस रोग जैसे इंट्रासेल्युलर एकत्रीकरण रोग।[4][36] ये उम्र की शुरुआत अपक्षयी रोग मिसफॉल्ड प्रोटीन के एकत्रीकरण से अघुलनशील, बाह्य समुच्चय और / या इंट्रासेल्युलर समावेशन में क्रॉस-β एमाइलॉयड महीन रेशा सहित जुड़े हुए हैं। यह पूरी तरह से स्पष्ट नहीं है कि समुच्चय कारण हैं या केवल प्रोटीन होमियोस्टेसिस के नुकसान का एक प्रतिबिंब है, संश्लेषण, वलन, एकत्रीकरण और प्रोटीन टर्नओवर के बीच संतुलन। हाल ही में यूरोपीय दवाई एजेंसी ने ट्रान्सथायरेटिन एमाइलॉयड रोगों के उपचार के लिए टैफिमिडिस या विंडाकेल (टेट्रामेरिक ट्रांसथायरेटिन का एक काइनेटिक स्टेबलाइजर) के उपयोग को मंजूरी दी है। इससे पता चलता है कि अमाइलॉइड फाइब्रिल गठन की प्रक्रिया (और स्वयं तंतु नहीं) मानव अमाइलॉइड रोगों में पोस्ट-माइटोटिक ऊतक के अध: पतन का कारण बनती है।[37] वलन और फंक्शन के बजाय मिसफॉल्डिंग और अत्यधिक गिरावट से ऐन्टीट्रिप्सिन से जुड़े वातस्फीति, सिस्टिक फाइब्रोसिस और लाइसोसोमल भंडारण रोग जैसे कई प्रोटियोंपैथी रोग हो जाते हैं, जहां फंक्शन की हानि विकार की उत्पत्ति है। जबकि प्रोटीन रिप्लेसमेंट थेरेपी का उपयोग ऐतिहासिक रूप से बाद के विकारों को ठीक करने के लिए किया गया है, एक उभरता हुआ दृष्टिकोण फार्मास्युटिकल चैपरोन का उपयोग उत्परिवर्तित प्रोटीन को फोल्ड करने के लिए उन्हें कार्यात्मक बनाने के लिए है।
प्रोटीन वलन का अध्ययन करने के लिए प्रायोगिक तकनीकें
जबकि प्रोटीन वलन के बारे में फी मान विश्लेषण के माध्यम से अनुमान लगाया जा सकता है, सामान्य रूप से, प्रोटीन वलन का अध्ययन करने के लिए प्रायोगिक तकनीकें प्रोटीन के संतुलन खुल रहा है या वलन पर निर्भर करती हैं और मानक गैर-क्रिस्टलोग्राफिक तकनीकों का उपयोग करके गठनात्मक परिवर्तनों का अवलोकन करती हैं।
एक्स-रे क्रिस्टलोग्राफी

एक्स-रे क्रिस्टलोग्राफी एक मुड़े हुए प्रोटीन के त्रि-आयामी विन्यास को समझने के प्रयास के लिए अधिक कुशल और महत्वपूर्ण तरीकों में से एक है।Cite error: Closing </ref> missing for <ref> tag[38] विकृतीकरण एक रासायनिक अणु (यूरिया, गनीडिनियम हाइड्रोक्लोराइड), तापमान, पीएच, दबाव, आदि हो सकता है। अलग-अलग लेकिन असतत प्रोटीन राज्यों के बीच संतुलन, यानी मूल राज्य, मध्यवर्ती राज्य, प्रकट राज्य, विकृतीकरण मूल्य पर निर्भर करता है; इसलिए, उनके संतुलन मिश्रण का वैश्विक प्रतिदीप्ति संकेत भी इस मान पर निर्भर करता है। इस प्रकार एक वैश्विक प्रोटीन संकेत को विकृतीकरण मूल्य से संबंधित एक प्रोफ़ाइल प्राप्त करता है। संतुलन के प्रकट होने की रूपरेखा किसी को प्रकट होने के मध्यवर्ती का पता लगाने और पहचानने में सक्षम कर सकती है।[39][40] ऐसे प्रोफाइल से ट्रिमर और संभावित टेट्रामर्स तक होमोमेरिक या हेटेरोमेरिक प्रोटीन के लिए प्रकट होने वाले संतुलन को चिह्नित करने वाले थर्मोडायनामिक पैरामीटर प्राप्त करने के लिए ह्यूजेस बेडौले द्वारा सामान्य समीकरण विकसित किए गए हैं।[41]प्रतिदीप्ति स्पेक्ट्रोस्कोपी को प्रोटीन वलन कैनेटीक्स को मापने के लिए रुके हुए प्रवाह जैसे तेजी से मिश्रण उपकरणों के साथ जोड़ा जा सकता है,[42] एक शेवरॉन प्लॉट उत्पन्न करें और एक Phi मान विश्लेषण प्राप्त करें।
वृत्ताकार द्वैतवाद
प्रोटीन वलन का अध्ययन करने के लिए परिपत्र द्विवर्णता सबसे सामान्य और बुनियादी उपकरणों में से एक है। वृत्ताकार द्वैतवाद स्पेक्ट्रोस्कोपी वृत्ताकार ध्रुवीकरण के अवशोषण को मापता है। प्रोटीन में, अल्फा हेलिक्स और बीटा शीट्स जैसी संरचनाएं चिरल होती हैं, और इस प्रकार इस तरह के प्रकाश को अवशोषित करती हैं। इस प्रकाश का अवशोषण प्रोटीन पहनावा की वलन की डिग्री के मार्कर के रूप में कार्य करता है। इस तकनीक का उपयोग विकृतीकरण एकाग्रता या तापमान के एक समारोह के रूप में इस अवशोषण में परिवर्तन को मापकर प्रोटीन के संतुलन को मापने के लिए किया गया है। एक डिनाट्यूरेंट मेल्ट अनवलन की थर्मोडायनामिक मुक्त ऊर्जा के साथ-साथ प्रोटीन के एम वैल्यू, या डिनेचुरेंट डिपेंडेंस को मापता है। पिघला हुआ तापमान प्रोटीन के विकृतीकरण मध्यबिंदु (टीएम) को मापता है।[41]प्रतिदीप्ति स्पेक्ट्रोस्कोपी के लिए, सर्कुलर-डाइक्रोइज्म स्पेक्ट्रोस्कोपी को प्रोटीन वलन रासायनिक गतिकी को मापने और शेवरॉन प्लॉट उत्पन्न करने के लिए रुके हुए प्रवाह जैसे फास्ट-मिक्सिंग उपकरणों के साथ जोड़ा जा सकता है।
प्रोटीन का कंपन वृत्ताकार द्वैतवाद
प्रोटीन के लिए कंपन परिपत्र द्वैतवाद (वीसीडी) तकनीकों के हाल के विकास, वर्तमान में फूरियर ट्रांसफॉर्म (एफटी) उपकरणों को सम्मिलित करते हुए, बहुत बड़े प्रोटीन अणुओं के लिए भी समाधान में प्रोटीन अनुरूपता निर्धारित करने के लिए शक्तिशाली साधन प्रदान करते हैं। प्रोटीन के ऐसे वीसीडी अध्ययनों को प्रोटीन क्रिस्टल के लिए एक्स-रे विवर्तन डेटा, भारी पानी में प्रोटीन समाधान के लिए एफटी आईआर डेटा (डी) के साथ जोड़ा जा सकता है।2ओ), या क्वांटम रसायन।
प्रोटीन परमाणु चुंबकीय अनुनाद स्पेक्ट्रोस्कोपी
प्रोटीन परमाणु चुंबकीय अनुनाद (NMR) केंद्रित प्रोटीन के नमूनों के माध्यम से चुंबक क्षेत्र को प्रेरित करके प्रोटीन संरचनात्मक डेटा एकत्र करने में सक्षम है। एनएमआर में, रासायनिक वातावरण के आधार पर, कुछ नाभिक विशिष्ट रेडियो-आवृत्तियों को अवशोषित करेंगे।[43][44] क्योंकि प्रोटीन संरचनात्मक परिवर्तन ns से ms तक के समय के पैमाने पर संचालित होते हैं, NMR विशेष रूप से ps से s के समयमानों में मध्यवर्ती संरचनाओं का अध्ययन करने के लिए सुसज्जित है।[45] प्रोटीन संरचना और गैर-वलन प्रोटीन संरचनात्मक परिवर्तनों का अध्ययन करने के लिए कुछ मुख्य तकनीकों में द्वि-आयामी परमाणु चुंबकीय अनुनाद स्पेक्ट्रोस्कोपी, द्वि-आयामी परमाणु चुंबकीय अनुनाद स्पेक्ट्रोस्कोपी, हेटेरोन्यूक्लियर सिंगल क्वांटम सुसंगतता स्पेक्ट्रोस्कोपी, आराम (NMR) (T1 और T2), और सम्मिलित हैं। परमाणु ओवरहॉसर प्रभाव।[43]एनओई विशेष रूप से उपयोगी है क्योंकि चुंबकीय स्थानान्तरण को स्थानिक रूप से समीपस्थ हाइड्रोजन्स के बीच देखा जा सकता है।[43]अलग-अलग एनएमआर प्रयोगों में टाइमस्केल संवेदनशीलता की अलग-अलग डिग्री होती है जो विभिन्न प्रोटीन संरचनात्मक परिवर्तनों के लिए उपयुक्त होती हैं। एनओई बॉन्ड कंपन या साइड चेन रोटेशन उठा सकता है, हालांकि, एनओई प्रोटीन वलन लेने के लिए बहुत संवेदनशील है क्योंकि यह बड़े पैमाने पर होता है।[45]
क्योंकि प्रोटीन वलन लगभग 50 से 3000 s में हो जाती है−1 सीपीएमजी रिलैक्सेशन डिस्पर्शन और चुंबकीयकरण स्थानांतरण वलन के एनएमआर विश्लेषण की कुछ प्राथमिक तकनीकें बन गई हैं।[44]इसके अलावा, प्रोटीन वलन परिदृश्य में उत्साहित मध्यवर्ती राज्यों को उजागर करने के लिए दोनों तकनीकों का उपयोग किया जाता है।[46] ऐसा करने के लिए, CPMG रिलैक्सेशन फैलाव स्पिन गूंज घटना का लाभ उठाता है। यह तकनीक लक्षित नाभिक को 90 पल्स के बाद एक या अधिक 180 दालों के बाद उजागर करती है।[47] न्यूक्लियर रिफोकस के रूप में, एक व्यापक वितरण इंगित करता है कि लक्ष्य न्यूक्लियर एक मध्यवर्ती उत्तेजित अवस्था में सम्मिलित है। रिलैक्सेशन डिस्पर्सन प्लॉट्स को देखकर डेटा उत्साहित और जमीन के बीच ऊष्मप्रवैगिकी और कैनेटीक्स पर जानकारी एकत्र करता है।[47][46]संतृप्ति स्थानांतरण जमीनी अवस्था से संकेत में परिवर्तन को मापता है क्योंकि उत्साहित अवस्थाएँ परेशान हो जाती हैं। यह एक विशेष नाभिक की उत्तेजित अवस्था को संतृप्त करने के लिए कमजोर रेडियो फ्रीक्वेंसी विकिरण का उपयोग करता है जो इसकी संतृप्ति को जमीनी अवस्था में स्थानांतरित करता है।[44]यह संकेत जमीनी अवस्था के चुंबकत्व (और संकेत) को कम करके बढ़ाया जाता है।[44][46]
NMR में मुख्य सीमाएँ यह हैं कि 25 kDa से बड़े प्रोटीन के साथ इसका विभेदन कम हो जाता है और यह एक्स-रे क्रिस्टलोग्राफी जितना विस्तृत नहीं है।[44]इसके अतिरिक्त, प्रोटीन एनएमआर विश्लेषण काफी कठिन है और एक ही एनएमआर स्पेक्ट्रम से कई समाधान प्रस्तावित कर सकता है।[43]
पेशीशोषी पार्श्व काठिन्य में सम्मिलित प्रोटीन SOD1 की वलन पर केंद्रित एक अध्ययन में, उत्साहित मध्यवर्ती का विश्राम फैलाव और संतृप्ति हस्तांतरण के साथ अध्ययन किया गया।[48] SOD1 को पहले कई बीमारी पैदा करने वाले म्यूटेंट से जोड़ा गया था, जिन्हें प्रोटीन एकत्रीकरण में सम्मिलित माना गया था, हालांकि तंत्र अभी भी अज्ञात था। आराम फैलाव और संतृप्ति हस्तांतरण प्रयोगों का उपयोग करके कई उत्साहित मध्यवर्ती राज्यों को SOD1 म्यूटेंट में मिसफॉल्डिंग का पर्दाफाश किया गया था।[48]
दोहरे ध्रुवीकरण इंटरफेरोमेट्री
दोहरी ध्रुवीकरण इंटरफेरोमेट्री आणविक परतों के ऑप्टिकल गुणों को मापने के लिए एक सवलन-आधारित तकनीक है। जब प्रोटीन वलन की विशेषता के लिए उपयोग किया जाता है, तो यह उप-एंग्स्ट्रॉम रिज़ॉल्यूशन पर वास्तविक समय में प्रोटीन के एक मोनोलेयर के समग्र आकार और इसके घनत्व को निर्धारित करके प्रोटीन की संरचना को मापता है।[49] हालांकि प्रोटीन वलन की कैनेटीक्स का रीयल-टाइम माप उन प्रक्रियाओं तक सीमित है जो ~10 Hz से धीमी होती हैं। वृत्ताकार द्वैतवाद के समान, वलन के लिए उत्तेजना एक विकृतिकारक या तापमान हो सकता है।
उच्च समय संकल्प के साथ वलन का अध्ययन
तेजी से, समयबद्ध तकनीकों के विकास से हाल के वर्षों में प्रोटीन वलन का अध्ययन बहुत उन्नत हुआ है। प्रयोगकर्ता तेजी से अनफोल्डेड प्रोटीन के नमूने की वलन को ट्रिगर करते हैं और परिणामी प्रोटीन गतिकी का निरीक्षण करते हैं। तेजी से उपयोग की जाने वाली तकनीकों में न्यूट्रॉन प्रकीर्णन सम्मिलित है,[50] समाधान, फोटोकैमिकल विधियों और तापमान कूद का अल्ट्राफास्ट मिश्रण। इन तकनीकों के विकास में योगदान देने वाले कई वैज्ञानिकों में जेरेमी कुक, हेनरिक रोडर, हैरी ग्रे (केमिस्ट), मार्टिन ग्रुबेले, ब्रायन डायर, विलियम ईटन, शीना रेडफोर्ड, क्रिस डॉब्सन, एलन फ़र्श, बेंग्ट नोल्टिंग और लार्स कोनेरमैन सम्मिलित हैं।
प्रोटियोलिसिस
प्रोटियोलिसिस का नियमित रूप से समाधान स्थितियों की एक विस्तृत श्रृंखला (जैसे तेज़ समानांतर प्रोटियोलिसिस (FASTpp)) के वलनत सामने आए अंश की जांच के लिए उपयोग किया जाता है।[51][52]
एकल-अणु बल स्पेक्ट्रोस्कोपी
ऑप्टिकल चिमटी और एएफएम जैसी एकल अणु तकनीकों का उपयोग अलग-अलग प्रोटीनों के प्रोटीन वलन तंत्र के साथ-साथ चैपरोन वाले प्रोटीनों को समझने के लिए किया गया है।[53] ऑप्टिकल चिमटी का उपयोग एकल प्रोटीन अणुओं को उनके सी- और एन-टर्मिनी से खींचने के लिए किया गया है और उन्हें बाद के रीवलन के अध्ययन की अनुमति देने के लिए प्रकट किया गया है।[54] तकनीक एकल-अणु स्तर पर वलन दरों को मापने की अनुमति देती है; उदाहरण के लिए, ऑप्टिकल चिमटी को हाल ही में रक्त जमावट में सम्मिलित प्रोटीनों को वलन और खोलने का अध्ययन करने के लिए लागू किया गया है। वॉन विलेब्रांड कारक (vWF) रक्त के थक्के बनने की प्रक्रिया में आवश्यक भूमिका वाला एक प्रोटीन है। इसने खोजा - एकल अणु ऑप्टिकल चिमटी माप का उपयोग करके - कि कैल्शियम-बाउंड वीडब्ल्यूएफ रक्त में कतरनी बल संवेदक के रूप में कार्य करता है। कतरनी बल vWF के A2 डोमेन को प्रकट करने की ओर ले जाता है, जिसकी रिफॉल्डिंग दर कैल्शियम की उपस्थिति में नाटकीय रूप से बढ़ जाती है।[55] हाल ही में, यह भी दिखाया गया था कि साधारण src SH3 डोमेन बल के वलनत कई अनवलन पाथवे तक पहुँचता है।[56]
बायोटिन पेंटिंग
बायोटिन पेंटिंग (अन) मुड़े हुए प्रोटीन के स्थिति-विशिष्ट सेलुलर स्नैपशॉट को सक्षम करती है। बायोटिन 'पेंटिंग' अनुमानित आंतरिक रूप से अव्यवस्थित प्रोटीन के प्रति पूर्वाग्रह दिखाती है।[57]
प्रोटीन वलन का कम्प्यूटेशनल अध्ययन
प्रोटीन वलन के कम्प्यूटेशनल अध्ययन में प्रोटीन स्थिरता, कैनेटीक्स और संरचना की भविष्यवाणी से संबंधित तीन मुख्य पहलू सम्मिलित हैं। 2013 की समीक्षा में प्रोटीन वलन के लिए उपलब्ध कम्प्यूटेशनल विधियों का सारांश दिया गया है।
[58]
लेविंथल का विरोधाभास
1969 में, साइरस लेविंथल ने नोट किया कि, एक अनफोल्डेड पॉलीपेप्टाइड श्रृंखला में स्वतंत्रता की बहुत बड़ी संख्या के कारण, अणु में खगोलीय संख्या में संभावित अनुरूपता होती है। 3 का अनुमान300 या 10143 उनके एक पेपर में बनाया गया था।[59] लेविंथल का विरोधाभास अवलोकन के आधार पर एक विचार प्रयोग है कि यदि प्रोटीन को सभी संभावित अनुरूपताओं के अनुक्रमिक नमूने से फोल्ड किया गया था, तो ऐसा करने में एक खगोलीय समय लगेगा, भले ही अनुरूपताओं को तीव्र दर (नैनोसेकंड पर) पर नमूना किया गया हो। या पीकोसैकन्ड स्केल)।[60] इस अवलोकन के आधार पर कि प्रोटीन इससे कहीं अधिक तेजी से मुड़ता है, लेविंथल ने तब प्रस्तावित किया कि एक यादृच्छिक रूपात्मक खोज नहीं होती है, और इसलिए प्रोटीन को मेटा-स्थिर प्रतिक्रिया मध्यवर्ती की एक श्रृंखला के माध्यम से मोड़ना चाहिए।
प्रोटीन वलन का ऊर्जा परिदृश्य
वलन के दौरान प्रोटीन के विन्यास स्थान (भौतिकी)भौतिकी) को ऊर्जा परिदृश्य के रूप में देखा जा सकता है। जोसेफ ब्रिंगल्सन और पीटर वोलिनेस के अनुसार, प्रोटीन न्यूनतम हताशा के सिद्धांत का पालन करते हैं, जिसका अर्थ है कि स्वाभाविक रूप से विकसित प्रोटीन ने अपने वलन ऊर्जा परिदृश्य को अनुकूलित किया है,[61] और उस प्रकृति ने अमीनो अम्ल अनुक्रमों को चुना है ताकि प्रोटीन की मुड़ी हुई अवस्था पर्याप्त रूप से स्थिर हो। इसके अलावा, मुड़े हुए राज्य का अधिग्रहण पर्याप्त रूप से तेज प्रक्रिया बनना था। भले ही प्रकृति ने प्रोटीन में हताशा के स्तर को कम कर दिया है, लेकिन इसका कुछ अंश अब तक बना हुआ है जैसा कि प्रोटीन के ऊर्जा परिदृश्य में स्थानीय मिनिमा की उपस्थिति में देखा जा सकता है।
इन क्रमिक रूप से चयनित अनुक्रमों का एक परिणाम यह है कि प्रोटीन को आम तौर पर विश्व स्तर पर फ़नल किए गए ऊर्जा परिदृश्य (जोस ओनुचिक द्वारा गढ़ा गया एक शब्द) माना जाता है।[62] जो मुख्य रूप से मूल राज्य की ओर निर्देशित हैं। यह वलन कीप लैंडस्केप प्रोटीन को किसी एक तंत्र तक सीमित होने के बजाय किसी भी बड़ी संख्या में रास्ते और मध्यवर्ती के माध्यम से मूल राज्य में वलन की अनुमति देता है। सिद्धांत जाली प्रोटीन और प्रायोगिक अध्ययन दोनों द्वारा समर्थित है,[61]और इसका उपयोग प्रोटीन संरचना भविष्यवाणी और प्रोटीन डिजाइन के तरीकों में सुधार के लिए किया गया है।[61]लेवलिंग फ्री-एनर्जी लैंडस्केप द्वारा प्रोटीन वलन का विवरण थर्मोडायनामिक्स के दूसरे कानून के अनुरूप भी है।[63] भौतिक रूप से, अधिकतम, सैडल पॉइंट्स, मिनिमा और फ़नल के साथ दृश्यमान क्षमता या कुल ऊर्जा सवलनों के संदर्भ में परिदृश्य के बारे में सोचना, बल्कि भौगोलिक परिदृश्य की तरह, शायद थोड़ा भ्रामक है। प्रासंगिक विवरण वास्तव में एक उच्च-आयामी चरण स्थान है जिसमें कई गुना अधिक जटिल टोपोलॉजिकल रूप ले सकते हैं।Cite error: Closing </ref> missing for <ref> tag प्रत्येक पथ के थर्मोडायनामिक अनुकूलता के आधार पर अलग-अलग रास्तों में उपयोग की अलग-अलग आवृत्तियाँ हो सकती हैं। इसका मतलब यह है कि यदि एक मार्ग दूसरे की तुलना में अधिक ऊष्मप्रवैगिक रूप से अनुकूल पाया जाता है, तो यह मूल संरचना की खोज में अधिक बार उपयोग किए जाने की संभावना है।[64]जैसे ही प्रोटीन मुड़ना प्रारम्भ करता है और इसके विभिन्न अनुरूपताएं ग्रहण करता है, यह हमेशा पहले की तुलना में अधिक ऊष्मागतिक रूप से अनुकूल संरचना की तलाश करता है और इस प्रकार ऊर्जा फ़नल के माध्यम से जारी रहता है। माध्यमिक संरचनाओं का निर्माण प्रोटीन के भीतर बढ़ी हुई स्थिरता का एक मजबूत संकेत है, और पॉलीपेप्टाइड रीढ़ की हड्डी द्वारा ग्रहण किए गए माध्यमिक संरचनाओं का केवल एक संयोजन सबसे कम ऊर्जा होगा और इसलिए प्रोटीन की मूल स्थिति में मौजूद होगा।[64]पॉलीपेप्टाइड के मुड़ने के बाद बनने वाली पहली संरचनाओं में अल्फा हेलिकॉप्टर और बीटा मोड़ हैं, जहां अल्फा हेलिकॉप्टर 100 नैनोसेकंड और बीटा 1 माइक्रोसेकंड में बदल सकते हैं।Cite error: Closing </ref> missing for <ref> tag निहित सॉल्वेंट मॉडल और छाता नमूनाकरण का उपयोग करके पहले संतुलन वलन सिमुलेशन किया गया था।[65] कम्प्यूटेशनल लागत के कारण, स्पष्ट पानी के साथ आरंभिक एमडी वलन सिमुलेशन पेप्टाइड्स और बहुत छोटे प्रोटीन तक सीमित हैं।[66][67] बड़े प्रोटीनों के एमडी सिमुलेशन प्रयोगात्मक संरचना की गतिशीलता या इसके उच्च तापमान के सामने आने तक ही सीमित रहते हैं। लंबे समय तक वलन करने की प्रक्रिया (लगभग 1 मिलीसेकंड से अधिक), जैसे छोटे आकार के प्रोटीन (लगभग 50 अवशेष) या बड़े की वलन, मोटे-दानेदार मॉडलिंग | मोटे-दानेदार मॉडल का उपयोग करके पहुँचा जा सकता है।[68][69][70]
कई बड़े पैमाने पर कम्प्यूटेशनल प्रोजेक्ट, जैसे Rosetta@home,[71] वलन @ घर[72] और मोड़ना,[73] लक्ष्य प्रोटीन वलन।
एंटोन (कंप्यूटर) पर लंबे निरंतर-प्रक्षेपवक्र सिमुलेशन का प्रदर्शन किया गया है, जो कि कस्टम ASICs और इंटरकनेक्ट्स के आसपास डीई शॉ रिसर्च द्वारा डिजाइन और निर्मित एक व्यापक समानांतर सुपरकंप्यूटर है। एंटोन का उपयोग करके किए गए सिमुलेशन का सबसे लंबा प्रकाशित परिणाम 355 K पर NTL9 का 2.936 मिलीसेकंड सिमुलेशन है।[74]
यह भी देखें
- शेवरॉन प्लॉट
- विकृतीकरण मध्यबिंदु
- डाउनहिल तह
- तह (रसायन विज्ञान)
- फी वैल्यू एनालिसिस
- प्रोटीन की संभावित ऊर्जा
- प्रोटीन गतिकी
- प्रोटीन मिसफॉल्डिंग चक्रीय प्रवर्धन
- प्रोटीन संरचना भविष्यवाणी सॉफ्टवेयर
- प्रोटिओपैथी
- समय-समाधान मास स्पेक्ट्रोमेट्री
संदर्भ
- ↑ Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walters P (2002). "The Shape and Structure of Proteins". कोशिका का आणविक जीवविज्ञान; चौथा संस्करण. New York and London: Garland Science. ISBN 978-0-8153-3218-3.
- ↑ Anfinsen CB (July 1972). "प्रोटीन संरचना का निर्माण और स्थिरीकरण". The Biochemical Journal. 128 (4): 737–49. doi:10.1042/bj1280737. PMC 1173893. PMID 4565129.
- ↑ Berg JM, Tymoczko JL, Stryer L (2002). "3. Protein Structure and Function". जीव रसायन. San Francisco: W. H. Freeman. ISBN 978-0-7167-4684-3.
- ↑ 4.0 4.1 Selkoe DJ (December 2003). "तह प्रोटीन घातक तरीके से". Nature. 426 (6968): 900–4. Bibcode:2003Natur.426..900S. doi:10.1038/nature02264. PMID 14685251. S2CID 6451881.
- ↑ Alberts B, Bray D, Hopkin K, Johnson A, Lewis J, Raff M, Roberts K, Walter P (2010). "Protein Structure and Function". आवश्यक कोशिका जीव विज्ञान (Third ed.). New York, NY: Garland Science. pp. 120–70. ISBN 978-0-8153-4454-4.
- ↑ Kim PS, Baldwin RL (1990). "छोटे प्रोटीनों की वलन अभिक्रियाओं में मध्यवर्ती होता है". Annual Review of Biochemistry. 59: 631–60. doi:10.1146/annurev.bi.59.070190.003215. PMID 2197986.
- ↑ Jackson SE (1998). "छोटे एकल-डोमेन प्रोटीन कैसे मोड़ते हैं?". Folding & Design. 3 (4): R81-91. doi:10.1016/S1359-0278(98)00033-9. PMID 9710577.
- ↑ Kubelka J, Hofrichter J, Eaton WA (February 2004). "प्रोटीन तह 'गति सीमा'". Current Opinion in Structural Biology. 14 (1): 76–88. doi:10.1016/j.sbi.2004.01.013. PMID 15102453.
- ↑ Scalvini, Barbara; Sheikhhassani, Vahid; Mashaghi, Alireza (2021). "प्रोटीन फोल्डिंग के टोपोलॉजिकल सिद्धांत". Physical Chemistry Chemical Physics. 23 (37): 21316–21328. Bibcode:2021PCCP...2321316S. doi:10.1039/D1CP03390E. hdl:1887/3277889. PMID 34545868. S2CID 237583577.
- ↑ Anfinsen CB (July 1973). "सिद्धांत जो प्रोटीन श्रृंखलाओं की तह को नियंत्रित करते हैं". Science. 181 (4096): 223–30. Bibcode:1973Sci...181..223A. doi:10.1126/science.181.4096.223. PMID 4124164.
- ↑ 11.0 11.1 11.2 11.3 11.4 11.5 11.6 11.7 Voet D, Voet JG, Pratt CW (2016). जैव रसायन के सिद्धांत (Fifth ed.). Wiley. ISBN 978-1-118-91840-1.
- ↑ Rose GD, Fleming PJ, Banavar JR, Maritan A (November 2006). "प्रोटीन फोल्डिंग का एक बैकबोन-आधारित सिद्धांत". Proceedings of the National Academy of Sciences of the United States of America. 103 (45): 16623–33. Bibcode:2006PNAS..10316623R. CiteSeerX 10.1.1.630.5487. doi:10.1073/pnas.0606843103. PMC 1636505. PMID 17075053.
- ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedFersht_1999 - ↑ "प्रोटीन संरचना". Scitable. Nature Education. Retrieved 2016-11-26.
- ↑ Pratt C, Cornely K (2004). "Thermodynamics". आवश्यक जैव रसायन. Wiley. ISBN 978-0-471-39387-0. Retrieved 2016-11-26.
- ↑ Zhang G, Ignatova Z (February 2011). "नवजात श्रृंखला के जन्म पर तह: सह-अनुवादिक तह के साथ अनुवाद का समन्वय". Current Opinion in Structural Biology. 21 (1): 25–31. doi:10.1016/j.sbi.2010.10.008. PMID 21111607.
- ↑ van den Berg B, Wain R, Dobson CM, Ellis RJ (August 2000). "मैक्रोमोलेक्युलर क्राउडिंग प्रोटीन रिफॉल्डिंग कैनेटीक्स को परेशान करता है: सेल के अंदर फोल्डिंग के लिए निहितार्थ". The EMBO Journal. 19 (15): 3870–5. doi:10.1093/emboj/19.15.3870. PMC 306593. PMID 10921869.
- ↑ Al-Karadaghi S. "प्रोटीन संरचना में मरोड़ कोण और रामचंद्रन प्लॉट". www.proteinstructures.com. Retrieved 2016-11-26.
- ↑ Pace CN, Shirley BA, McNutt M, Gajiwala K (January 1996). "प्रोटीन के गठनात्मक स्थिरता में योगदान देने वाली शक्तियाँ". FASEB Journal. 10 (1): 75–83. doi:10.1096/fasebj.10.1.8566551. PMID 8566551. S2CID 20021399.
- ↑ Cui D, Ou S, Patel S (December 2014). "हाइड्रोफोबिक प्रभावों के माध्यम से मध्यस्थता किए गए प्रोटीन-प्रोटीन इंटरैक्शन की भविष्यवाणी के लिए प्रोटीन-फैले हुए पानी के नेटवर्क और निहितार्थ". Proteins. 82 (12): 3312–26. doi:10.1002/prot.24683. PMID 25204743. S2CID 27113763.
- ↑ Tanford C (June 1978). "हाइड्रोफोबिक प्रभाव और जीवित पदार्थ का संगठन". Science. 200 (4345): 1012–8. Bibcode:1978Sci...200.1012T. doi:10.1126/science.653353. PMID 653353.
- ↑ Deechongkit S, Nguyen H, Powers ET, Dawson PE, Gruebele M, Kelly JW (July 2004). "बीटा-शीट फोल्डिंग एनर्जेटिक्स के लिए बैकबोन हाइड्रोजन बॉन्डिंग का प्रसंग-निर्भर योगदान". Nature. 430 (6995): 101–5. Bibcode:2004Natur.430..101D. doi:10.1038/nature02611. PMID 15229605. S2CID 4315026.
- ↑ Irbäck A, Sandelin E (November 2000). "प्रोटीन श्रृंखलाओं में हाइड्रोफोबिसिटी सहसंबंध पर". Biophysical Journal. 79 (5): 2252–8. arXiv:cond-mat/0010390. Bibcode:2000BpJ....79.2252I. doi:10.1016/S0006-3495(00)76472-1. PMC 1301114. PMID 11053106.
- ↑ Irbäck A, Peterson C, Potthast F (September 1996). "प्रोटीन श्रृंखलाओं में गैर-यादृच्छिक हाइड्रोफोबिसिटी संरचनाओं के लिए साक्ष्य". Proceedings of the National Academy of Sciences of the United States of America. 93 (18): 9533–8. arXiv:chem-ph/9512004. Bibcode:1996PNAS...93.9533I. doi:10.1073/pnas.93.18.9533. PMC 38463. PMID 8790365.
- ↑ Wilson BA, Foy SG, Neme R, Masel J (June 2017). "डे नोवो जीन जन्म". Nature Ecology & Evolution. 1 (6): 0146–146. doi:10.1038/s41559-017-0146. PMC 5476217. PMID 28642936.
- ↑ Willis S, Masel J (September 2018). "जीन जन्म ओवरलैपिंग जीन द्वारा एन्कोडेड संरचनात्मक विकार में योगदान देता है". Genetics. 210 (1): 303–313. doi:10.1534/genetics.118.301249. PMC 6116962. PMID 30026186.
- ↑ Foy SG, Wilson BA, Bertram J, Cordes MH, Masel J (April 2019). "एकत्रीकरण परिहार रणनीति में बदलाव प्रोटीन विकास के लिए एक दीर्घकालिक दिशा को चिह्नित करता है". Genetics. 211 (4): 1345–1355. doi:10.1534/genetics.118.301719. PMC 6456324. PMID 30692195.
- ↑ 28.0 28.1 Cite error: Invalid
<ref>tag; no text was provided for refs namedDobson_2003 - ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedHartl_1996 - ↑ Shortle D (January 1996). "विकृत अवस्था (तह समीकरण का दूसरा भाग) और प्रोटीन स्थिरता में इसकी भूमिका". FASEB Journal. 10 (1): 27–34. doi:10.1096/fasebj.10.1.8566543. PMID 8566543. S2CID 24066207.
- ↑ Lee S, Tsai FT (2005). "प्रोटीन गुणवत्ता नियंत्रण में आणविक संरक्षक". Journal of Biochemistry and Molecular Biology. 38 (3): 259–65. doi:10.5483/BMBRep.2005.38.3.259. PMID 15943899.</रेफरी> अघुलनशील अनाकार समुच्चय में वर्षा (रसायन विज्ञान) के जोखिम को रोकने के लिए यह कार्य महत्वपूर्ण है। प्रोटीन विकृतीकरण या देशी राज्य के विघटन में शामिल बाहरी कारकों में तापमान, बाहरी क्षेत्र (विद्युत, चुंबकीय), शामिल हैं। रेफरी नाम = ओजेडा >Ojeda-May P, Garcia ME (July 2010). "देशी बीटा-शीट प्रोटीन संरूपण का विद्युत क्षेत्र-संचालित व्यवधान और एक हेलिक्स-संरचना का निर्माण". Biophysical Journal. 99 (2): 595–9. Bibcode:2010BpJ....99..595O. doi:10.1016/j.bpj.2010.04.040. PMC 2905109. PMID 20643079.</ रेफ> आणविक भीड़, रेफरी नाम = बर्ग >van den Berg B, Ellis RJ, Dobson CM (December 1999). "प्रोटीन फोल्डिंग और एग्रीगेशन पर मैक्रोमोलेक्यूलर क्राउडिंग का प्रभाव". The EMBO Journal. 18 (24): 6927–33. doi:10.1093/emboj/18.24.6927. PMC 1171756. PMID 10601015.
- ↑ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (August 2008). "122 डिग्री सेल्सियस पर सेल प्रसार और उच्च दबाव की खेती के तहत हाइपरथर्मोफिलिक मेथनोजेन द्वारा समस्थानिक रूप से भारी सीएच4 उत्पादन". Proceedings of the National Academy of Sciences of the United States of America. 105 (31): 10949–54. Bibcode:2008PNAS..10510949T. doi:10.1073/pnas.0712334105. PMC 2490668. PMID 18664583.
- ↑ 33.0 33.1 Marusich EI, Kurochkina LP, Mesyanzhinov VV. Chaperones in bacteriophage T4 assembly. Biochemistry (Mosc). 1998;63(4):399-406
- ↑ Porter, Lauren L.; Looger, Loren L. (5 June 2018). "मौजूदा फोल्ड-स्विचिंग प्रोटीन व्यापक हैं". Proceedings of the National Academy of Sciences. 115 (23): 5968–5973. doi:10.1073/pnas.1800168115. PMC 6003340. PMID 29784778.
- ↑ Cite error: Invalid
<ref>tag; no text was provided for refs namedChaudhuri_2006 - ↑ Chiti F, Dobson CM (2006). "प्रोटीन मिसफॉल्डिंग, कार्यात्मक अमाइलॉइड और मानव रोग". Annual Review of Biochemistry. 75: 333–66. doi:10.1146/annurev.biochem.75.101304.123901. PMID 16756495.
- ↑ Johnson SM, Wiseman RL, Sekijima Y, Green NS, Adamski-Werner SL, Kelly JW (December 2005). "प्रोटीन मिसफॉल्डिंग बीमारियों को ठीक करने की रणनीति के रूप में नेटिव स्टेट काइनेटिक स्थिरीकरण: ट्रान्सथायरेटिन एमाइलॉयडोज पर ध्यान केंद्रित". Accounts of Chemical Research. 38 (12): 911–21. doi:10.1021/ar020073i. PMID 16359163.
- ↑ Monsellier E, Bedouelle H (September 2005). "प्रतिदीप्ति अधिकतम तरंग दैर्ध्य के साथ मॉनिटर किए गए संतुलन से प्रोटीन स्थिरता की मात्रात्मक माप". Protein Engineering, Design & Selection. 18 (9): 445–56. doi:10.1093/protein/gzi046. PMID 16087653.
- ↑ Park YC, Bedouelle H (July 1998). "बेसिलस स्टीरोथर्मोफिलस से डिमेरिक टायरोसिल-टीआरएनए सिंथेटेस एक मोनोमेरिक इंटरमीडिएट के माध्यम से सामने आता है। संतुलन शर्तों के तहत एक मात्रात्मक विश्लेषण". The Journal of Biological Chemistry. 273 (29): 18052–9. doi:10.1074/jbc.273.29.18052. PMID 9660761.
- ↑ Ould-Abeih MB, Petit-Topin I, Zidane N, Baron B, Bedouelle H (June 2012). "मल्टीपल फोल्डिंग स्टेट्स एंड डिसऑर्डर ऑफ राइबोसोमल प्रोटीन एसए, लैमिनिन, एंटीकार्सिनोजेन्स और रोगजनकों के लिए एक झिल्ली रिसेप्टर". Biochemistry. 51 (24): 4807–21. doi:10.1021/bi300335r. PMID 22640394.
- ↑ 41.0 41.1 Cite error: Invalid
<ref>tag; no text was provided for refs namedpmid=26607240 - ↑ Royer CA (May 2006). "प्रतिदीप्ति के साथ प्रोटीन फोल्डिंग और कंफॉर्मल ट्रांजिशन की जांच करना". Chemical Reviews. 106 (5): 1769–84. doi:10.1021/cr0404390. PMID 16683754.
- ↑ 43.0 43.1 43.2 43.3 Wüthrich K (December 1990). "एनएमआर स्पेक्ट्रोस्कोपी द्वारा समाधान में प्रोटीन संरचना निर्धारण". The Journal of Biological Chemistry. 265 (36): 22059–62. doi:10.1016/S0021-9258(18)45665-7. PMID 2266107.
- ↑ 44.0 44.1 44.2 44.3 44.4 Zhuravleva A, Korzhnev DM (May 2017). "एनएमआर द्वारा प्रोटीन तह". Progress in Nuclear Magnetic Resonance Spectroscopy. 100: 52–77. doi:10.1016/j.pnmrs.2016.10.002. PMID 28552172.
- ↑ 45.0 45.1 Ortega G, Pons M, Millet O (2013-01-01). Karabencheva-Christova T (ed.). "एनएमआर स्पेक्ट्रोस्कोपी द्वारा अध्ययन के रूप में प्रोटीन कार्यात्मक गतिशीलता कई समयमानों में". Advances in Protein Chemistry and Structural Biology. Dynamics of Proteins and Nucleic Acids. Academic Press. 92: 219–51. doi:10.1016/b978-0-12-411636-8.00006-7. ISBN 9780124116368. PMID 23954103.
- ↑ 46.0 46.1 46.2 Vallurupalli P, Bouvignies G, Kay LE (May 2012). "एक प्रमुख राज्य रचना के साथ धीमी गति से "अदृश्य" उत्साहित प्रोटीन राज्यों का अध्ययन". Journal of the American Chemical Society. 134 (19): 8148–61. doi:10.1021/ja3001419. PMID 22554188.
- ↑ 47.0 47.1 Neudecker P, Lundström P, Kay LE (March 2009). "प्रोटीन तह के विस्तृत अध्ययन के लिए एक उपकरण के रूप में विश्राम फैलाव एनएमआर स्पेक्ट्रोस्कोपी". Biophysical Journal. 96 (6): 2045–54. Bibcode:2009BpJ....96.2045N. doi:10.1016/j.bpj.2008.12.3907. PMC 2717354. PMID 19289032.
- ↑ 48.0 48.1 Sekhar A, Rumfeldt JA, Broom HR, Doyle CM, Sobering RE, Meiering EM, Kay LE (November 2016). "NMR स्पेक्ट्रोस्कोपी द्वारा SOD1 के ALS रोग म्यूटेंट के मुक्त ऊर्जा परिदृश्य की जांच करना". Proceedings of the National Academy of Sciences of the United States of America. 113 (45): E6939–E6945. doi:10.1073/pnas.1611418113. PMC 5111666. PMID 27791136.
- ↑ Cross GH, Freeman NJ, Swann MJ (2008). "Dual Polarization Interferometry: A Real-Time Optical Technique for Measuring (Bio)molecular Orientation, Structure and Function at the Solid/Liquid Interface". बायोसेंसर और बायोचिप्स की हैंडबुक. doi:10.1002/9780470061565.hbb055. ISBN 978-0-470-01905-4.
- ↑ Bu Z, Cook J, Callaway DJ (September 2001). "देशी और विकृत अल्फा-लैक्टलबुमिन में गतिशील शासन और सहसंबद्ध संरचनात्मक गतिशीलता". Journal of Molecular Biology. 312 (4): 865–73. doi:10.1006/jmbi.2001.5006. PMID 11575938.
- ↑ Minde DP, Maurice MM, Rüdiger SG (2012). "तेजी से प्रोटियोलिसिस परख, FASTpp द्वारा लाइसेट्स में बायोफिजिकल प्रोटीन स्थिरता का निर्धारण". PLOS ONE. 7 (10): e46147. Bibcode:2012PLoSO...746147M. doi:10.1371/journal.pone.0046147. PMC 3463568. PMID 23056252.
- ↑ Park C, Marqusee S (March 2005). "पल्स प्रोटियोलिसिस: प्रोटीन स्थिरता और लिगैंड बाइंडिंग के मात्रात्मक निर्धारण के लिए एक सरल विधि". Nature Methods. 2 (3): 207–12. doi:10.1038/nmeth740. PMID 15782190. S2CID 21364478.
- ↑ Mashaghi A, Kramer G, Lamb DC, Mayer MP, Tans SJ (January 2014). "एकल-अणु स्तर पर संरक्षिका क्रिया". Chemical Reviews. 114 (1): 660–76. doi:10.1021/cr400326k. PMID 24001118.
- ↑ Jagannathan B, Marqusee S (November 2013). "बल के तहत प्रोटीन फोल्डिंग और अनफोल्डिंग". Biopolymers. 99 (11): 860–9. doi:10.1002/bip.22321. PMC 4065244. PMID 23784721.
- ↑ Jakobi AJ, Mashaghi A, Tans SJ, Huizinga EG (July 2011). "कैल्शियम वॉन विलेब्रांड कारक A2 डोमेन द्वारा बल संवेदन को नियंत्रित करता है". Nature Communications. 2: 385. Bibcode:2011NatCo...2..385J. doi:10.1038/ncomms1385. PMC 3144584. PMID 21750539.
- ↑ Jagannathan B, Elms PJ, Bustamante C, Marqusee S (October 2012). "एक प्रोटीन के अनिसोट्रोपिक मैकेनिकल अनफोल्डिंग पाथवे में एक बल-प्रेरित स्विच का प्रत्यक्ष अवलोकन". Proceedings of the National Academy of Sciences of the United States of America. 109 (44): 17820–5. Bibcode:2012PNAS..10917820J. doi:10.1073/pnas.1201800109. PMC 3497811. PMID 22949695.
- ↑ Minde DP, Ramakrishna M, Lilley KS (2018). "प्रॉक्सिमिटी लेबलिंग द्वारा बायोटिनाइलेशन अनफोल्डेड प्रोटीन का समर्थन करता है". bioRxiv. doi:10.1101/274761.
- ↑ Compiani M, Capriotti E (December 2013). "प्रोटीन तह के लिए कम्प्यूटेशनल और सैद्धांतिक तरीके". Biochemistry. 52 (48): 8601–24. doi:10.1021/bi4001529. PMID 24187909.
- ↑ "स्ट्रक्चरल बायोकैमिस्ट्री/प्रोटीन/प्रोटीन फोल्डिंग - विकिपुस्तकें, खुली दुनिया के लिए खुली किताबें". en.wikibooks.org. Retrieved 2016-11-05.
- ↑ Levinthal C (1968). "क्या प्रोटीन फोल्डिंग के रास्ते हैं?" (PDF). Journal de Chimie Physique et de Physico-Chimie Biologique. 65: 44–45. Bibcode:1968JCP....65...44L. doi:10.1051/jcp/1968650044. Archived from the original (PDF) on 2009-09-02.
- ↑ 61.0 61.1 61.2 Bryngelson JD, Onuchic JN, Socci ND, Wolynes PG (March 1995). "फ़नल, पाथवे और प्रोटीन फोल्डिंग का ऊर्जा परिदृश्य: एक संश्लेषण". Proteins. 21 (3): 167–95. arXiv:chem-ph/9411008. doi:10.1002/prot.340210302. PMID 7784423. S2CID 13838095.
- ↑ Leopold PE, Montal M, Onuchic JN (September 1992). "प्रोटीन तह फ़नल: अनुक्रम-संरचना संबंध के लिए एक गतिज दृष्टिकोण". Proceedings of the National Academy of Sciences of the United States of America. 89 (18): 8721–5. Bibcode:1992PNAS...89.8721L. doi:10.1073/pnas.89.18.8721. PMC 49992. PMID 1528885.
- ↑ Sharma V, Kaila VR, Annila A (2009). "एक विकासवादी प्रक्रिया के रूप में प्रोटीन तह". Physica A: Statistical Mechanics and Its Applications. 388 (6): 851–62. Bibcode:2009PhyA..388..851S. doi:10.1016/j.physa.2008.12.004.
- ↑ 64.0 64.1 Cite error: Invalid
<ref>tag; no text was provided for refs namedDill_2012 - ↑ Schaefer M, Bartels C, Karplus M (December 1998). "समाधान अनुरूपता और संरचित पेप्टाइड्स के ऊष्मप्रवैगिकी: आणविक गतिकी सिमुलेशन एक अंतर्निहित सॉल्वेशन मॉडल के साथ". Journal of Molecular Biology. 284 (3): 835–48. doi:10.1006/jmbi.1998.2172. PMID 9826519.
- ↑ Jones D. "फ्रैगमेंट-आधारित प्रोटीन फोल्डिंग सिमुलेशन". University College London.
- ↑ "प्रोटीन की तह" (by Molecular Dynamics).
- ↑ Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A (July 2016). "मोटे अनाज वाले प्रोटीन मॉडल और उनके अनुप्रयोग". Chemical Reviews. 116 (14): 7898–936. doi:10.1021/acs.chemrev.6b00163. PMID 27333362.
- ↑ Kmiecik S, Kolinski A (July 2007). "रिड्यूस्ड-स्पेस मॉडलिंग द्वारा प्रोटीन-फोल्डिंग पाथवे का लक्षण वर्णन". Proceedings of the National Academy of Sciences of the United States of America. 104 (30): 12330–5. Bibcode:2007PNAS..10412330K. doi:10.1073/pnas.0702265104. PMC 1941469. PMID 17636132.
- ↑ Adhikari AN, Freed KF, Sosnick TR (October 2012). "अनुक्रमिक स्थिरीकरण के सिद्धांत का उपयोग करके प्रोटीन फोल्डिंग पाथवे और संरचना की नए सिरे से भविष्यवाणी". Proceedings of the National Academy of Sciences of the United States of America. 109 (43): 17442–7. Bibcode:2012PNAS..10917442A. doi:10.1073/pnas.1209000109. PMC 3491489. PMID 23045636.
- ↑ Rosetta@Home
- ↑ Folding@Home
- ↑ FoldIt - Folding Protein Game
- ↑ Lindorff-Larsen K, Piana S, Dror RO, Shaw DE (October 2011). "कितनी तेजी से फोल्डिंग प्रोटीन फोल्ड होता है". Science. 334 (6055): 517–20. Bibcode:2011Sci...334..517L. doi:10.1126/science.1208351. PMID 22034434. S2CID 27988268.
इस पेज में लापता आंतरिक लिंक की सूची
- शारीरिक प्रक्रिया
- कलफ़
- बीमारी
- प्रतिरक्षा तंत्र
- जलाना
- खाना बनाना
- संपर्क आदेश
- अल्फा हेलिक्स
- बैकबोन चेन
- वैन डेर वाल बल
- गठनात्मक एंट्रॉपी
- हाइड्रोफोबिक पतन
- तापीय धारिता
- फिर से जन्म जीन
- लाइव
- ग्रोस
- टैफामाइड्स
- फुरियर रूपांतरण
- चरण की समस्या
- आंशिक प्राप्ति
- प्रवाह बंद कर दिया
- गोलाकार ध्रुवीकरण
- एक्स - रे विवर्तन
- नाभिकीय चुबकीय अनुनाद
- आराम (एनएमआर)
- प्रोटीन रचना
- एलन फ़र्स्ट
- तापमान में उछाल
- तेजी से समानांतर प्रोटियोलिसिस (FASTpp)
- जालीदार प्रोटीन
- मोटे दाने वाली मॉडलिंग