बायोइनफॉरमैटिक्स

जैव सूचना विज्ञान (/ˌbaɪ.oʊˌɪnfərˈmætɪks/ (![]() listen)) जीव विज्ञान का अंतःविषय क्षेत्र है जो जीवविज्ञान डेटा को समझने के लिए तरीकों और सॉफ्टवेयर टूल विकसित करता है, खासकर जब डेटा समूह बड़े और समष्टि होते हैं। तब जैव सूचना विज्ञान जैविक डेटा का विश्लेषण और व्याख्या करने के लिए जीव विज्ञान, रसायन विज्ञान, भौतिकी, कंप्यूटर विज्ञान, कंप्यूटर प्रोग्रामिंग, सूचना इंजिनियरिंग (क्षेत्र), गणित और सांख्यिकी का उपयोग करता है। डेटा के विश्लेषण और व्याख्या की पश्चात् की प्रक्रिया को कम्प्यूटेशनल जीवविज्ञान कहा जाता है।
listen)) जीव विज्ञान का अंतःविषय क्षेत्र है जो जीवविज्ञान डेटा को समझने के लिए तरीकों और सॉफ्टवेयर टूल विकसित करता है, खासकर जब डेटा समूह बड़े और समष्टि होते हैं। तब जैव सूचना विज्ञान जैविक डेटा का विश्लेषण और व्याख्या करने के लिए जीव विज्ञान, रसायन विज्ञान, भौतिकी, कंप्यूटर विज्ञान, कंप्यूटर प्रोग्रामिंग, सूचना इंजिनियरिंग (क्षेत्र), गणित और सांख्यिकी का उपयोग करता है। डेटा के विश्लेषण और व्याख्या की पश्चात् की प्रक्रिया को कम्प्यूटेशनल जीवविज्ञान कहा जाता है।
जैविक प्रश्नों के सिलिको में विश्लेषण के लिए कम्प्यूटेशनल, सांख्यिकीय और कंप्यूटर प्रोग्रामिंग तकनीकों का उपयोग किया गया है। उनमें पुन: उपयोग की जाने वाली विशिष्ट विश्लेषण पाइप लाइनें सम्मिलित हैं, विशेष रूप से जीनोमिक्स के क्षेत्र में, जैसे कि जीन और एकल न्यूक्लियोटाइड बहुरूपता (एकल-न्यूक्लियोटाइड बहुरूपता) की पहचान करके। इन पाइपलाइनों का उपयोग बीमारी के आनुवंशिक आधार, अद्वितीय अनुकूलन, वांछनीय गुणों (विशेषकर कृषि प्रजातियों में), या जनसंख्या के मध्य अंतर को उत्तम ढंग से समझने के लिए किया जाता है। जैव सूचना विज्ञान में प्रोटिओमिक्स भी सम्मिलित है, जो न्यूक्लिक एसिड और प्रोटीन अनुक्रमों के अंदर संगठनात्मक सिद्धांतों को समझने की कोशिश करता है।[1]
छवि और सिग्नल प्रोसेसिंग बड़ी मात्रा में कच्चे डेटा से उपयोगी परिणाम निकालने की अनुमति देती है। आनुवंशिकी के क्षेत्र में, यह जीनोम और उनके देखे गए उत्परिवर्तनों को अनुक्रमित करने में और व्याख्या करने में सहायता करता है। जैव सूचना विज्ञान में जैविक साहित्य का पाठ खनन और जैविक डेटा को व्यवस्थित और क्वेरी करने के लिए जैविक और जीन ओन्टोलॉजी (सूचना विज्ञान) का विकास सम्मिलित है। यह जीन और प्रोटीन अभिव्यक्ति और विनियमन के विश्लेषण में भी भूमिका निभाता है। जैव सूचना विज्ञान उपकरण आनुवंशिक और जीनोमिक डेटा की तुलना, विश्लेषण और व्याख्या करने और सामान्यतः आणविक जीव विज्ञान के विकासवादी पहलुओं को समझने में सहायता करते हैं। अधिक एकीकृत स्तर पर, यह उन जैविक मार्गों और नेटवर्कों का विश्लेषण और सूचीबद्ध करने में मदद करता है जो कि प्रणाली जीव विज्ञान का महत्वपूर्ण हिस्सा हैं। संरचनात्मक जीव विज्ञान में, यह डीएनए,आरएनए,[2][3] तथा प्रोटीन[4] के साथ ही जैव-आणविक अंतःक्रियाओं केअनुकरण और मॉडलिंग में सहायता करता है,[2] [5][6][7][8]
इतिहास
जैव सूचना विज्ञान शब्द की पहली परिभाषा 1970 में पॉलियन होगेवेग और बेन हेस्पर द्वारा जैविक प्रणालियों में सूचना प्रक्रियाओं के अध्ययन को संदर्भित करने के लिए गढ़ी गई थी।[9][10][11][12][13] इस परिभाषा ने जैव सूचना विज्ञान को जैव रसायन (जैविक प्रणालियों में रासायनिक प्रक्रियाओं का अध्ययन) के समानांतर क्षेत्र के रूप में रखा।[10]
जैव सूचना विज्ञान और कम्प्यूटेशनल जीव विज्ञान में जैविक डेटा, विशेष रूप से डीएनए, आरएनए और प्रोटीन अनुक्रमों का विश्लेषण सम्मिलित था। 1990 के दशक के मध्य में जैव सूचना विज्ञान के क्षेत्र में विस्फोटक वृद्धि का अनुभव हुआ, जो मुख्य रूप से मानव जीनोम परियोजना और डीएनए अनुक्रमण प्रौद्योगिकी में तेजी से प्रगति से प्रेरित था।
सार्थक जानकारी उत्पन्न करने के लिए जैविक डेटा का विश्लेषण करने में सॉफ्टवेयर प्रोग्राम लिखना और चलाना सम्मिलित है जो ग्राफ सिद्धांत, कृत्रिम बुद्धिमत्ता, सॉफ्ट कंप्यूटिंग, डेटा खनन, मूर्ति प्रोद्योगिकी और कंप्यूटर सिमुलेशन से कलन विधि का उपयोग करते हैं। बदले में एल्गोरिदम सैद्धांतिक आधारों जैसे कि भिन्न गणित, नियंत्रण सिद्धांत, प्रणालीसिद्धांत, सूचना सिद्धांत और सांख्यिकी पर निर्भर करते हैं।
अनुक्रम
मानव जीनोम परियोजना के पूरा होने के पश्चात् से गति और निवेश में जबरदस्त प्रगति हुई है, कुछ प्रयोगशालाएँ हर साल 100,000 बिलियन से अधिक आधारों को अनुक्रमित करने में सक्षम हैं, और पूर्ण जीनोम को 1,000 डॉलर या उससे कम में अनुक्रमित किया जा सकता है।[14] 1950 के दशक की प्रारंभिक में फ्रेडरिक सिंगर द्वारा इंसुलिन का अनुक्रम निर्धारित करने के पश्चात् प्रोटीन अनुक्रम उपलब्ध होने पर कंप्यूटर आणविक जीव विज्ञान में आवश्यक हो गए। अनेक अनुक्रमों की मैन्युअल रूप से तुलना करना अव्यावहारिक निकला। मार्गरेट ओकले डेहॉफ़, इस क्षेत्र में अग्रणी,[15] पहले प्रोटीन अनुक्रम डेटाबेस में से को संकलित किया गया था जिसे प्रारंभ में पुस्तकों के रूप में प्रकाशित किया गया था[16] साथ ही अनुक्रम संरेखण और आणविक विकास के तरीके।[17] जैव सूचना विज्ञान में और प्रारंभिक योगदानकर्ता एल्विन ए. काबट थे, जिन्होंने 1980 और 1991 के मध्य ताई ते वू के साथ ऑनलाइन जारी एंटीबॉडी अनुक्रमों की व्यापक मात्रा के साथ 1970 में जैविक अनुक्रम विश्लेषण का बीड़ा उठाया था।[18]
1970 के दशक में, डीएनए अनुक्रमण के लिए नई तकनीकों को बैक्टीरियोफेज MS2 और øX174 पर प्रयुक्त किया गया था, और विस्तारित न्यूक्लियोटाइड अनुक्रमों को फिर सूचनात्मक और सांख्यिकीय एल्गोरिदम के साथ पार्स किया गया था। इन अध्ययनों से पता चला है कि कोडिंग सेगमेंट और ट्रिपलेट कोड जैसी प्रसिद्ध विशेषताएं सीधे सांख्यिकीय विश्लेषणों में सामने आती हैं और इस अवधारणा का प्रमाण थीं कि जैव सूचना विज्ञान व्यावहारिक होगा।[19][20]
लक्ष्य
यह अध्ययन करने के लिए कि विभिन्न रोग स्थितियों में सामान्य सेलुलर गतिविधियाँ कैसे बदल जाती हैं, इन गतिविधियों की व्यापक तस्वीर बनाने के लिए कच्चे जैविक डेटा को जोड़ा जाना चाहिए। इसलिए, जैव सूचना विज्ञान का क्षेत्र इस तरह विकसित हुआ है कि अभी सबसे महत्वपूर्ण कार्य में विभिन्न प्रकार के डेटा का विश्लेषण और व्याख्या सम्मिलित है। इसमें न्यूक्लियोटाइड और अमीनो एसिड अनुक्रम, प्रोटीन डोमेन और प्रोटीन संरचनाएं भी सम्मिलित हैं।[21] जैव सूचना विज्ञान और कम्प्यूटेशनल जीव विज्ञान के महत्वपूर्ण उप-विषयों में सम्मिलित हैं:
- विभिन्न प्रकार की सूचनाओं तक कुशलतापूर्वक पहुंच, प्रबंधन और उपयोग करने के लिए कंप्यूटर प्रोग्राम का विकास और कार्यान्वयन।
- बड़े डेटा समूह के सदस्यों के मध्य संबंधों का आकलन करने के लिए नए गणितीय एल्गोरिदम और सांख्यिकीय उपायों का विकास। उदाहरण के लिए, अनुक्रम के अंदर जीन का पता लगाने, प्रोटीन संरचना और/या कार्य की भविष्यवाणी करने और संबंधित अनुक्रमों के परिवारों में प्रोटीन अनुक्रमों का क्लस्टर विश्लेषण करने के तरीके हैं।
जैव सूचना विज्ञान का प्राथमिक लक्ष्य जैविक प्रक्रियाओं की समझ को बढ़ाना है। जो चीज़ इसे अन्य दृष्टिकोणों से भिन्न करती है, वह इस लक्ष्य को प्राप्त करने के लिए कम्प्यूटेशनल रूप से गहन तकनीकों को विकसित करने और प्रयुक्त करने पर केंद्रित है। उदाहरणों में सम्मिलित हैं: पैटर्न पहचान, डेटा माइनिंग, यंत्र अधिगम एल्गोरिदम और जैविक डेटा विज़ुअलाइज़ेशन। क्षेत्र में प्रमुख अनुसंधान प्रयासों में अनुक्रम संरेखण, जीन खोज, जीनोम असेंबली, दवा डिजाइन, दवा खोज, प्रोटीन संरचनात्मक संरेखण, प्रोटीन संरचना भविष्यवाणी, जीन अभिव्यक्ति की भविष्यवाणी और प्रोटीन-प्रोटीन इंटरैक्शन, जीनोम-वाइड एसोसिएशन अध्ययन, विकास का मॉडलिंग सम्मिलित है। और सेलुलर मॉडल|कोशिका विभाजन/माइटोसिस।
जैव सूचना विज्ञान में जैविक डेटा के प्रबंधन और विश्लेषण से उत्पन्न होने वाली औपचारिक और व्यावहारिक समस्याओं को हल करने के लिए डेटाबेस, एल्गोरिदम, कम्प्यूटेशनल और सांख्यिकीय तकनीकों में सिद्धांत का निर्माण और उन्नति सम्मिलित है।
पिछले कुछ दशकों में, जीनोमिक और अन्य आणविक अनुसंधान प्रौद्योगिकियों में तेजी से विकास और सूचना प्रौद्योगिकियों में विकास ने आणविक जीव विज्ञान से संबंधित जबरदस्त मात्रा में जानकारी का उत्पादन किया है। जैव सूचना विज्ञान इन गणितीय और कंप्यूटिंग दृष्टिकोणों को दिया गया नाम है जिनका उपयोग जैविक प्रक्रियाओं की समझ बढ़ाने के लिए किया जाता है।
जैव सूचना विज्ञान में सामान्य गतिविधियों में डीएनए और प्रोटीन अनुक्रमों का मानचित्रण और विश्लेषण करना सम्मिलित है। तथा उनकी तुलना करने के लिए डीएनए और प्रोटीन अनुक्रमों को संरेखित करना भी सम्मिलित है। प्रोटीन संरचनाओं के 3-डी मॉडल बनाना और देखना भी सम्मिलित है।
अनुक्रम विश्लेषण
चूँकि 1977 में बैक्टीरियो के फेज Φ-X174 का अनुक्रमण किया गया था,[22] हजारों जीवों के डीएनए अनुक्रमों को डीकोड किया गया है और डेटाबेस में संग्रहीत किया गया है। इस अनुक्रम जानकारी का विश्लेषण उन जीनों को निर्धारित करने के लिए किया जाता है जो कि प्रोटीन, आरएनए जीन, नियामक अनुक्रम, संरचनात्मक रूपांकनों और दोहराव वाले अनुक्रमों को एन्कोड करते हैं। किसी प्रजाति के अंदर या विभिन्न प्रजातियों के मध्य जीन की तुलना प्रोटीन कार्यों, या प्रजातियों के मध्य संबंधों (फाइलोजेनेटिक पेड़ों के निर्माण के लिए आणविक प्रणाली विज्ञान का उपयोग) के मध्य समानता दिखा सकती है। डेटा की बढ़ती मात्रा के साथ, डीएनए अनुक्रमों का मैन्युअल रूप से विश्लेषण करना बहुत पहले अव्यावहारिक हो गया था। ब्लास्ट जैसे कंप्यूटर प्रोग्राम का उपयोग नियमित रूप से अनुक्रम खोजने के लिए किया जाता है - 2008 तक, 260,000 से अधिक जीवों से, जिनमें 190 बिलियन से अधिक न्यूक्लियोटाइड होते हैं।[23]
डीएनए अनुक्रमण
अनुक्रमों का विश्लेषण करने से पहले, उन्हें जेनबैंक जैसे डेटा स्टोरेज बैंक से प्राप्त किया जाता है। डीएनए अनुक्रमण अभी भी गैर-साधारण समस्या है क्योंकि कच्चा डेटा ध्वनि हो सकता है या अशक्त संकेतों से प्रभावित हो सकता है। डीएनए अनुक्रमण के विभिन्न प्रयोगात्मक दृष्टिकोणों के लिए आधार कॉलिंग के लिए एल्गोरिदम विकसित किए गए हैं।
अनुक्रम संयोजन
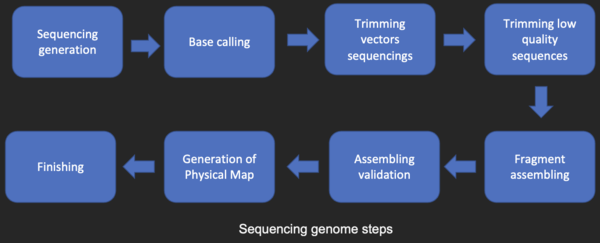
अधिकांश डीएनए अनुक्रमण तकनीकें अनुक्रम के छोटे टुकड़े उत्पन्न करती हैं जिन्हें पूर्ण जीन या जीनोम अनुक्रम प्राप्त करने के लिए इकट्ठा करने की आवश्यकता होती है। शॉटगन अनुक्रमण विधि (पहले जीवाणु जीनोम, हेमोफिलस इन्फ्लुएंजा को अनुक्रमित करने के लिए इंस्टीट्यूट फॉर जीनोमिक रिसर्च (टीआईजीआर) द्वारा उपयोग किया जाता है)[24] अनेक हजारों छोटे डीएनए टुकड़ों (अनुक्रमण विधि के आधार पर 35 से 900 न्यूक्लियोटाइड तक लंबे) के अनुक्रम उत्पन्न करता है। इन टुकड़ों के सिरे ओवरलैप होते हैं और, जब जीनोम असेंबली प्रोग्राम द्वारा ठीक से संरेखित किया जाता है, तब संपूर्ण जीनोम के पुनर्निर्माण के लिए उपयोग किया जा सकता है। शॉटगन अनुक्रमण से अनुक्रम डेटा तुरंत प्राप्त होता है, किन्तु बड़े जीनोम के लिए टुकड़ों को इकट्ठा करने का कार्य अधिक समष्टि हो सकता है। मानव जीनोम जितने बड़े जीनोम के लिए, टुकड़ों को इकट्ठा करने के लिए बड़े-मेमोरी, मल्टीप्रोसेसर कंप्यूटर पर अनेक दिनों का सीपीयू जितना समय लग सकता है, और परिणामी असेंबली में सामान्यतः अनेक अंतराल होते हैं जिन्हें पश्चात् में भरना होगा। शॉटगन अनुक्रमण लगभग सभी अनुक्रमित जीनोम के लिए पसंद की विधि है (श्रृंखला-समाप्ति या रासायनिक गिरावट विधियों के अतिरिक्त ), और जीनोम असेंबली एल्गोरिदम जैव सूचना विज्ञान अनुसंधान का महत्वपूर्ण क्षेत्र है।
जीनोम एनोटेशन
जीनोमिक्स में, जीनोम प्रोजेक्ट या जीनोम एनोटेशन अनुक्रमित डीएनए अनुक्रम में जीन और अन्य जैविक विशेषताओं के रुकने और प्रारंभ करने वाले क्षेत्रों को चिह्नित करने की प्रक्रिया को संदर्भित करता है। अनेकजीनोम इतने बड़े हैं कि उन्हें हाथ से एनोटेट नहीं किया जा सकता। चूंकि डीएनए अनुक्रमण की दर जीनोम एनोटेशन की दर से अधिक हो गई है, जीनोम एनोटेशन जैव सूचना विज्ञान में नई बाधा बन गया है।.
जीनोम एनोटेशन को तीन स्तरों में वर्गीकृत किया जा सकता है: न्यूक्लियोटाइड, प्रोटीन और प्रक्रिया स्तर।
जीन खोज न्यूक्लियोटाइड-स्तर एनोटेशन का मुख्य कथन है। समष्टि जीनोम के लिए, एब इनिटियो जीन भविष्यवाणी और व्यक्त अनुक्रम डेटाबेस और अन्य जीवों के साथ अनुक्रम तुलना का संयोजन सफल हो सकता है। न्यूक्लियोटाइड-स्तर एनोटेशन जीनोम के अन्य आनुवंशिक और भौतिक मानचित्रों के साथ जीनोम अनुक्रम के एकीकरण की भी अनुमति देता है।
प्रोटीन-स्तरीय एनोटेशन का मुख्य उद्देश्य जीनोम के प्रोटीन उत्पादों को कार्य प्रदर्शित करना है। इस प्रकार के एनोटेशन के लिए प्रोटीन अनुक्रमों और कार्यात्मक डोमेन और रूपांकनों के डेटाबेस का उपयोग किया जाता है। नए जीनोम अनुक्रम में अनुमानित प्रोटीनों में से लगभग आधे का कोई स्पष्ट कार्य नहीं होता है।
सेलुलर और जीव विज्ञान के संदर्भ में जीन और उनके उत्पादों के कार्य को समझना प्रक्रिया-स्तरीय एनोटेशन का लक्ष्य है। प्रक्रिया-स्तरीय एनोटेशन की बाधा विभिन्न मॉडल प्रणालियों द्वारा उपयोग किए जाने वाले शब्दों की असंगति रही है। जीन ओन्टोलॉजी कंसोर्टियम इस समस्या को हल करने में मदद कर रहा है।[25] व्यापक एनोटेशन प्रणाली का पहला विवरण 1995 में प्रकाशित हुआ था[24] इंस्टीट्यूट फॉर जीनोमिक रिसर्च द्वारा, जिसने मुक्त-जीवित जीव, जीवाणु हीमोफिलस इन्फ्लुएंजा के जीनोम का पहला पूर्ण अनुक्रमण और विश्लेषण किया।[24] प्रणाली प्रारंभिक कार्यात्मक कार्य करने के लिए सभी प्रोटीनों को एनकोडिंग करने वाले, आरएनए, राइबोसोमल आरएनए को स्थानांतरित करने वाले जीन की पहचान करता है। हेमोफिलस इन्फ्लुएंजा में प्रोटीन-कोडिंग जीन खोजने के लिए प्रशिक्षित जीनमार्क कार्यक्रम लगातार बदल रहा है और सुधार कर रहा है।
2003 में बंद होने के पश्चात् मानव जीनोम परियोजना ने जिन लक्ष्यों को प्राप्त करना छोड़ दिया था, उनका अनुसरण करते हुए, एनकोड परियोजना को राष्ट्रीय मानव जीनोम अनुसंधान संस्थान द्वारा विकसित किया गया था। यह परियोजना मानव जीनोम के कार्यात्मक तत्वों का सहयोगी डेटा संग्रह है जो अगली पीढ़ी के डीएनए-अनुक्रमण प्रौद्योगिकियों और जीनोमिक टाइलिंग सरणी का उपयोग करती है, प्रौद्योगिकियां नाटकीय रूप से कम प्रति-आधार निवेश पर स्वचालित रूप से बड़ी मात्रा में डेटा उत्पन्न करने में सक्षम हैं किन्तु समान के साथ स्पष्टता (आधार कॉल त्रुटि) और निष्ठा (असेंबली त्रुटि)।
जीन फलन भविष्यवाणी
जबकि जीनोम एनोटेशन मुख्य रूप से अनुक्रम समानता (और इस प्रकार होमोलॉजी (जीव विज्ञान)) पर आधारित है, अनुक्रमों के अन्य गुणों का उपयोग जीन के कार्य की भविष्यवाणी करने के लिए किया जा सकता है। वास्तव में, अधिकांश जीन फलन भविष्यवाणी विधियां प्रोटीन अनुक्रमों पर ध्यान केंद्रित करती हैं क्योंकि वह अधिक जानकारीपूर्ण और अधिक सुविधा संपन्न हैं। उदाहरण के लिए, हाइड्रोफोबिक एमिनो एसिड का वितरण प्रोटीन में ट्रांसमेम्ब्रेन डोमेन की भविष्यवाणी करता है। चूँकि, प्रोटीन फलन भविष्यवाणी बाहरी जानकारी जैसे जीन (या प्रोटीन) जीन अभिव्यक्ति डेटा, प्रोटीन संरचना, या प्रोटीन-प्रोटीन इंटरैक्शन|प्रोटीन-प्रोटीन इंटरैक्शन का भी उपयोग कर सकती है।[26]
कम्प्यूटेशनल विकासवादी जीवविज्ञान
विकासवादी जीवविज्ञान प्रजातियों की उत्पत्ति और वंश के साथ-साथ समय के साथ उनके परिवर्तन का अध्ययन है। सूचना विज्ञान (शैक्षणिक क्षेत्र) ने शोधकर्ताओं को निम्नलिखित में सक्षम बनाकर विकासवादी जीवविज्ञानियों की सहायता की है:
- केवल भौतिक वर्गीकरण या शारीरिक टिप्पणियों के अतिरिक्त , बड़ी संख्या में जीवों के डीएनए में परिवर्तन को मापकर उनके विकास का पता लगाएं,
- संपूर्ण जीनोम की तुलना करें, जो अधिक समष्टि विकासवादी घटनाओं, जैसे जीन दोहराव, क्षैतिज जीन स्थानांतरण और जीवाणु प्रजाति में महत्वपूर्ण कारकों की भविष्यवाणी के अध्ययन की अनुमति देता है,
- समय के साथ प्रणाली के परिणाम की भविष्यवाणी करने के लिए समष्टि कम्प्यूटेशनल जनसंख्या आनुवंशिकी मॉडल का निर्माण करें[27]
- बड़ी संख्या में प्रजातियों और जीवों पर जानकारी ट्रैक करें और साझा करें |
भविष्य का कार्य अभी और अधिक समष्टि विकासवादी वृक्ष के पुनर्निर्माण का प्रयास करता है।
तुलनात्मक जीनोमिक्स
तुलनात्मक जीनोम विश्लेषण का मूल विभिन्न जीवों में जीन होमोलॉजी (जीवविज्ञान) या ऑर्थोलॉजी विश्लेषण) या अन्य जीनोमिक विशेषताओं के मध्य पत्राचार की स्थापना है। इंटरजेनोमिक मानचित्र दो जीनोम के विचलन के लिए जिम्मेदार विकासवादी प्रक्रियाओं का पता लगाने के लिए बनाए जाते हैं। विभिन्न संगठनात्मक स्तरों पर कार्य करने वाली अनेक विकासवादी घटनाएं जीनोम विकास को आकार देती हैं। निम्नतम स्तर पर, बिंदु उत्परिवर्तन व्यक्तिगत न्यूक्लियोटाइड को प्रभावित करते हैं। उच्च स्तर पर, बड़े गुणसूत्र खंड दोहराव, पार्श्व स्थानांतरण, व्युत्क्रम, स्थानांतरण, विलोपन और सम्मिलन से गुजरते हैं।[28] संपूर्ण जीनोम संकरण, पॉलीप्लोइडाइजेशन और एंडोसिंबियोसिस की प्रक्रियाओं में सम्मिलित होते हैं जो तेजी से प्रजातिकरण की ओर ले जाते हैं। तथा जीनोम विकास की जटिलता गणितीय मॉडल और एल्गोरिदम के डेवलपर्स के लिए अनेक रोमांचक चुनौतियां प्रस्तुत करती है, जिनके पास पारसीमोनी मॉडल पर आधारित समस्याओं के लिए स्पष्ट, अनुमान, निश्चित पैरामीटर और सन्निकटन एल्गोरिदम से लेकर मार्कोव तक एल्गोरिथम, सांख्यिकीय और गणितीय तकनीकों के स्पेक्ट्रम का सहारा होता है। संभाव्य मॉडल पर आधारित समस्याओं के बायेसियन विश्लेषण के लिए श्रृंखला मोंटे कार्लो एल्गोरिदम।
इनमें से अनेक अध्ययन प्रोटीन परिवार को अनुक्रम निर्दिष्ट करने के लिए अनुक्रम समरूपता का पता लगाने पर आधारित हैं।[29]
पैन जीनोमिक्स
पैन जीनोमिक्स 2005 में टेटेलिन और मेदिनी द्वारा प्रस्तुत की गई अवधारणा है। पैन जीनोम विशेष मोनोफिली टैक्सोनोमिक समूह का संपूर्ण जीन भंडार है। चूँकि प्रारंभ में इसे किसी प्रजाति के निकट संबंधी उपभेदों पर प्रयुक्त किया गया था, किन्तु इसे जीनस, फ़ाइल में आदि जैसे बड़े संदर्भों पर भी प्रयुक्त किया जा सकता है। इसे दो भागों में विभाजित किया गया है: कोर जीनोम, अध्ययन के अनुसार सभी जीनोमों के लिए सामान्य जीन का समूह (अधिकांशतः जीवित रहने के लिए महत्वपूर्ण हाउसकीपिंग जीन), और डिस्पेंसेबल/लचीला जीनोम: जीन का समूह जो अध्ययन के अनुसार या कुछ जीनोम को छोड़कर सभी में उपस्तिथ नहीं है। जीवाणु प्रजातियों के पैन जीनोम को चिह्नित करने के लिए जैव सूचना विज्ञान उपकरण बीपीजीए का उपयोग किया जा सकता है।[30]
रोग की आनुवंशिकी
2013 तक, कुशल उच्च-थ्रूपुट अगली पीढ़ी की अनुक्रमण विधि का अस्तित्व अनेक भिन्न -भिन्न मानव विकारों के कारणों की पहचान करने की अनुमति देता है। ऑनलाइन मेंडेलियन इनहेरिटेंस इन मैन डेटाबेस में पहचाने गए 3,000 से अधिक विकारों के लिए सरल मेंडेलियन वंशानुक्रम देखा गया है, किन्तु समष्टि बीमारियाँ अधिक कठिन हैं। एसोसिएशन के अध्ययनों में अनेक व्यक्तिगत आनुवंशिक क्षेत्र पाए गए हैं जो व्यक्तिगत रूप से समष्टि बीमारियों (जैसे बांझपन,) से अशक्त रूप से जुड़े हुए हैं।[31] स्तन कैंसर[32] और [33] किसी कारण के अतिरिक्त ।[34][35] वर्तमान में निदान और उपचार के लिए जीन का उपयोग करने में अनेकचुनौतियाँ हैं, जैसे कि हम कैसे नहीं जानते कि कौन से जीन महत्वपूर्ण हैं, या एल्गोरिदम द्वारा प्रदान किए जाने वाले विकल्प कितने स्थिर हैं। [36]
जीनोम-वाइड एसोसिएशन अध्ययनों ने समष्टि बीमारियों और लक्षणों के लिए हजारों सामान्य आनुवंशिक वेरिएंट की सफलतापूर्वक पहचान की है; चूँकि, यह सामान्य रूप आनुवंशिकता के छोटे से अंश की ही व्याख्या करते हैं।[37] दुर्लभ कार्यात्मक संस्करण कुछ लुप्त आनुवंशिकता समस्या का कारण हो सकता है।[38] बड़े पैमाने पर संपूर्ण जीनोम अनुक्रमण अध्ययनों ने तेजी से लाखों संपूर्ण जीनोमों को अनुक्रमित किया है, और ऐसे अध्ययनों ने करोड़ों दुर्लभ कार्यात्मक प्रकारों की पहचान की है।[39] एसएनपी एनोटेशन आनुवंशिक वेरिएंट के प्रभाव या कार्य की भविष्यवाणी करता है और दुर्लभ कार्यात्मक वेरिएंट को प्राथमिकता देने में मदद करता है, और इन एनोटेशन को सम्मिलित करने से पूरे जीनोम अनुक्रमण अध्ययन के दुर्लभ वेरिएंट विश्लेषण के आनुवंशिक सहयोग की शक्ति को प्रभावी ढंग से बढ़ावा मिल सकता है।[40] संपूर्ण-जीनोम अनुक्रमण डेटा के लिए ऑल-इन-वन दुर्लभ वैरिएंट एसोसिएशन विश्लेषण प्रदान करने के लिए कुछ उपकरण विकसित किए गए हैं, जिसमें जीनोटाइप डेटा और उनके कार्यात्मक एनोटेशन, एसोसिएशन विश्लेषण, परिणाम सारांश और विज़ुअलाइज़ेशन का एकीकरण सम्मिलित है।[41][42] संपूर्ण जीनोम अनुक्रमण अध्ययन का मेटा-विश्लेषण समष्टि फेनोटाइप से जुड़े दुर्लभ वेरिएंट की खोज के लिए बड़े नमूना आकार एकत्र करने की समस्या का आकर्षक समाधान प्रदान करता है।[43]
कैंसर में उत्परिवर्तन का विश्लेषण
कैंसर में, प्रभावित कोशिकाओं के जीनोम को समष्टि या अप्रत्याशित तरीकों से पुनर्व्यवस्थित किया जाता है। कैंसर का कारण बनने वाले बिंदु उत्परिवर्तन की पहचान करने वाले एकल-न्यूक्लियोटाइड बहुरूपता सरणियों के अतिरिक्त, ऑलिगोन्यूक्लियोटाइड माइक्रोएरे का उपयोग क्रोमोसोमल लाभ और हानि (तुलनात्मक जीनोमिक संकरण कहा जाता है) की पहचान करने के लिए किया जा सकता है। यह पता लगाने की विधियाँ प्रति प्रयोग टेराबाइट डेटा उत्पन्न करती हैं। डेटा में अधिकांशतः अधिक परिवर्तनशीलता, या ध्वनि पाई जाता है, और इस प्रकार वास्तविक प्रतिलिपि संख्या भिन्नता परिवर्तनों का अनुमान लगाने के लिए छिपा हुआ मार्कोव मॉडल और परिवर्तन-बिंदु विश्लेषण विधियां विकसित की जा रही हैं।
निर्वासित में उत्परिवर्तन द्वारा कैंसर की पहचान करने के लिए दो महत्वपूर्ण सिद्धांतों का उपयोग किया जा सकता है। सबसे पहले, कैंसर जीन में संचित दैहिक उत्परिवर्तन की बीमारी है। दूसरा, कैंसर में ड्राइवर उत्परिवर्तन होते हैं जिन्हें यात्रियों से भिन्न करने की आवश्यकता होती है।[44]
जैव सूचना विज्ञान में और सुधार से जीनोम में कैंसर प्रेरित उत्परिवर्तन के विश्लेषण द्वारा कैंसर के प्रकारों को वर्गीकृत करने की अनुमति मिल सकती है। इसके अतिरिक्त, भविष्य में कैंसर के प्रतिरूपों के अनुक्रम से रोग बढ़ने पर रोगियों पर नज़र रखना संभव हो सकता है। अन्य प्रकार का डेटा जिसके लिए नवीन सूचना विज्ञान विकास की आवश्यकता होती है, वहअनेक ट्यूमर के मध्य बार-बार होने वाले घावों का विश्लेषण होता है ।[45]
जीन और प्रोटीन अभिव्यक्ति
जीन अभिव्यक्ति का विश्लेषण
डीएनए माइक्रोएरे, व्यक्त अनुक्रम टैग (ईएसटी) अनुक्रमण, जीन अभिव्यक्ति का क्रमिक विश्लेषण (एसएजीई) टैग अनुक्रमण, व्यापक समानांतर हस्ताक्षर अनुक्रमण (एमपीएसएस), आरएनए- सहित अनेक तकनीकों के साथ मैसेंजर आरएनए स्तरों को मापकर अनेक जीनों की जीन अभिव्यक्ति निर्धारित की जा सकती है। जिसे होल ट्रांस्क्रिप्टोम शॉटगन सीक्वेंसिंग (डब्ल्यूटीएसएस) या मल्टीप्लेक्स इन-सीटू हाइब्रिडाइजेशन के विभिन्न अनुप्रयोगों के रूप में भी जाना जाता है। यह सभी तकनीकें अत्यधिक ध्वनि-प्रवण हैं और यह जैविक माप में पूर्वाग्रह के अधीन हैं, और कम्प्यूटेशनल जीव विज्ञान में प्रमुख अनुसंधान क्षेत्र में उच्च-थ्रूपुट जीन अभिव्यक्ति अध्ययनों में ध्वनि से सिग्नल (सूचना सिद्धांत) को भिन्न करने के लिए सांख्यिकीय उपकरण विकसित करना सम्मिलित है।[46] इस तरह के अध्ययनों का उपयोग अधिकांशतः किसी विकार में सम्मिलित जीन को निर्धारित करने के लिए किया जाता है: कैंसर कोशिकाओं की विशेष जनसंख्या में अप-विनियमित और डाउन-विनियमित प्रतिलेखों को निर्धारित करने के लिए कैंसरग्रस्त उपकला कोशिकाओं के माइक्रोएरे डेटा की तुलना गैर-कैंसर कोशिकाओं के डेटा से की जा सकती है। .
प्रोटीन अभिव्यक्ति का विश्लेषण
प्रोटीन माइक्रोएरे और उच्च थ्रूपुट (एचटी) मास स्पेक्ट्रोमेट्री (एमएस) जैविक प्रतिरूप में उपस्तिथ प्रोटीन का स्नैपशॉट प्रदान कर सकते हैं। पूर्व दृष्टिकोण को एमआरएनए पर लक्षित माइक्रोएरे के समान समस्याओं का सामना करना पड़ता है, पश्चात् वाले में प्रोटीन अनुक्रम डेटाबेस से अनुमानित द्रव्यमान के विरुद्ध बड़ी मात्रा में बड़े पैमाने पर डेटा के मिलान की समस्या सम्मिलित होती है, और प्रत्येक प्रोटीन से अनेकअपूर्ण पेप्टाइड्स का पता चलने पर प्रतिरूपों का समष्टि सांख्यिकीय विश्लेषण होता है। ऊतक संदर्भ में सेलुलर प्रोटीन स्थानीयकरण को इम्युनोहिस्टोकैमिस्ट्री और ऊतक माइक्रोएरे के आधार पर स्थानिक डेटा के रूप में प्रदर्शित एफ़िनिटी प्रोटिओमिक्स के माध्यम से प्राप्त किया जा सकता है।[47]
नियमन का विश्लेषण
जीन अभिव्यक्ति का विनियमन समष्टि प्रक्रिया है जहां संकेत, जैसे कि हार्मोन जैसे बाह्य कोशिकीय संकेत, अंततः या अधिक प्रोटीन की गतिविधि में वृद्धि या कमी की ओर ले जाता है। इस प्रक्रिया में विभिन्न चरणों का पता लगाने के लिए जैव सूचना विज्ञान तकनीकों को प्रयुक्त किया गया है।
उदाहरण के लिए, जीन अभिव्यक्ति को जीनोम में आस-पास के तत्वों द्वारा नियंत्रित किया जा सकता है। प्रमोटर विश्लेषण में जीन के प्रोटीन-कोडिंग क्षेत्र के आसपास के डीएनए में अनुक्रम रूपांकनों की पहचान और अध्ययन सम्मिलित है। यह रूपांकन उस सीमा को प्रभावित करते हैं जिस सीमा तक उस क्षेत्र को एमआरएनए में स्थानांतरित किया जाता है। प्रवर्तक से दूर संवर्द्धक (आनुवांशिकी) तत्व त्रि-आयामी लूपिंग इंटरैक्शन के माध्यम से जीन अभिव्यक्ति को भी नियंत्रित कर सकते हैं। इन अंतःक्रियाओं को गुणसूत्र संरचना कैप्चर प्रयोगों के जैव सूचनात्मक विश्लेषण द्वारा निर्धारित किया जा सकता है।
अभिव्यक्ति डेटा का उपयोग जीन विनियमन का अनुमान लगाने के लिए किया जा सकता है: प्रत्येक राज्य में सम्मिलित जीन के बारे में परिकल्पना बनाने के लिए किसी जीव की विभिन्न अवस्थाओं से माइक्रोएरे डेटा की तुलना की जा सकती है। एकल-कोशिका जीव में, कोई कोशिका चक्र के चरणों की तुलना विभिन्न तनाव स्थितियों (गर्मी का झटका, भुखमरी, आदि) के साथ कर सकता है। फिर क्लस्टर विश्लेषण को अभिव्यक्ति डेटा पर प्रयुक्त किया जा सकता है जिससे कि यह निर्धारित किया जा सके कि कौन से जीन सह-व्यक्त हैं। उदाहरण के लिए, सह-व्यक्त जीन के अपस्ट्रीम क्षेत्रों (प्रमोटरों) को अधिक प्रतिनिधित्व वाले नियामक तत्वों के लिए खोजा जा सकता है। जीन क्लस्टरिंग में प्रयुक्त क्लस्टरिंग एल्गोरिदम के उदाहरण हैं k-कारण क्लस्टरिंग , सेल्फ-ऑर्गनाइजिंग मैप्स (एसओएम), पदानुक्रमित क्लस्टरिंग और सर्वसम्मति क्लस्टरिंग विधियां होती है ।
सेलुलर संगठन का विश्लेषण
कोशिकाओं के अंदर ऑर्गेनेल, जीन, प्रोटीन और अन्य घटकों के स्थान का विश्लेषण करने के लिए अनेक दृष्टिकोण विकसित किए गए हैं। तथा अनेक जैविक डेटाबेस में उपसेलुलर स्थानीयकरण को पकड़ने के लिए जीन ऑन्टोलॉजी श्रेणी, सेलुलर घटक को तैयार किया गया है।
माइक्रोस्कोपी और छवि विश्लेषण
सूक्ष्म चित्र ऑर्गेनेल के साथ-साथ अणुओं के स्थान की भी जानकारी देते हैं, जो रोगों में असामान्यताओं का स्रोत हो सकते हैं।
प्रोटीन स्थानीयकरण
प्रोटीन का स्थान खोजने से हमें यह अनुमान लगाने की अनुमति मिलती है कि वह क्या करते हैं। इसे प्रोटीन फलन भविष्यवाणी कहा जाता है। उदाहरण के लिए, यदि कोशिका नाभिक में प्रोटीन पाया जाता है तब यह जीन अभिव्यक्ति या आरएनए स्प्लिसिंग के विनियमन में सम्मिलित हो सकता है। इसके विपरीत, यदि माइटोकॉन्ड्रियन में प्रोटीन पाया जाता है, तब यह सेलुलर श्वसन या अन्य नामकरण में सम्मिलित हो सकता है। प्रोटीन उपसेलुलर स्थान डेटाबेस और भविष्यवाणी उपकरण सहित अच्छी तरह से विकसित प्रोटीन उपसेलुलर स्थानीयकरण भविष्यवाणी संसाधन उपलब्ध हैं।[48][49]
क्रोमेटिन का परमाणु संगठन
उच्च-थ्रूपुट क्रोमोसोम संरचना कैप्चर प्रयोगों से डेटा, जैसे कि हाई-सी (जीनोमिक विश्लेषण विधि ) | हाई-सी (प्रयोग) और चिया-पीईटी, क्रोमेटिन की त्रि-आयामी संरचना और परमाणु संगठन पर जानकारी प्रदान कर सकते हैं। इस क्षेत्र में जैव सूचनात्मक चुनौतियों में जीनोम को डोमेन में विभाजित करना सम्मिलित है, जैसे कि टोपोलॉजिकली एसोसिएटिंग डोमेन (टीएडी), जो त्रि-आयामी अंतरिक्ष में साथ व्यवस्थित होते हैं।[50]
संरचनात्मक जैव सूचना विज्ञान
प्रोटीन की संरचना का पता लगाना जैव सूचना विज्ञान का महत्वपूर्ण अनुप्रयोग है। प्रोटीन संरचना भविष्यवाणी का महत्वपूर्ण आकलन (सीएएसपी) विवर्त प्रतियोगिता है जहां विश्वभर के अनुसंधान समूह अज्ञात प्रोटीन मॉडल के मूल्यांकन के लिए प्रोटीन मॉडल प्रस्तुत करते हैं।[51][52]
अमीनो एसिड अनुक्रम
प्रोटीन के रैखिक अमीनो एसिड अनुक्रम को प्राथमिक संरचना कहा जाता है, इसे डीएनए जीन पर कोडन के अनुक्रम से आसानी से निर्धारित किया जा सकता है जो इसके लिए कोड करता है। अधिकांश प्रोटीनों में, प्राथमिक संरचना विशिष्ट रूप से अपने मूल वातावरण में प्रोटीन की 3-आयामी संरचना को निर्धारित करती है। अपवाद मूर्ख बोवाइनो को होने वाला रोग में सम्मिलित प्रियन है। यह संरचना प्रोटीन के कार्य से जुड़ी होती है। अतिरिक्त संरचनात्मक जानकारी में द्वितीयक संरचना, तृतीयक संरचना और चतुर्धातुक संरचना संरचना में सम्मिलित हैं। प्रोटीन के कार्य की भविष्यवाणी के लिए व्यवहार्य सामान्य समाधान विवर्त समस्या बनी हुई है। अभी तक अधिकांश प्रयास उन अनुमानों की ओर निर्देशित किए गए हैं जो अधिकांश समय तक काम करते हैं।
समरूपता
जैव सूचना विज्ञान की जीनोमिक शाखा में, जीन के कार्य की भविष्यवाणी करने के लिए होमोलॉजी का उपयोग किया जाता है: यदि जीन A का अनुक्रम, जिसका कार्य ज्ञात है, जीन B के अनुक्रम के अनुरूप है, जिसका कार्य अज्ञात है, तब कोई यह अनुमान लगा सकता है कि B हो सकता है तथा A के कार्य को साझा करें। संरचनात्मक जैव सूचना विज्ञान में, होमोलॉजी का उपयोग यह निर्धारित करने के लिए किया जाता है कि प्रोटीन के कौन से हिस्से संरचना निर्माण और अन्य प्रोटीन के साथ बातचीत में महत्वपूर्ण हैं। होमोलॉजी मॉडलिंग का उपयोग उपस्थित समजात प्रोटीन से अज्ञात प्रोटीन की संरचना की भविष्यवाणी करने के लिए किया जाता है।
इसका उदाहरण मनुष्यों में हीमोग्लोबिन और फलियों में हीमोग्लोबिन (लेगहीमोग्लोबिन ) है, जो ही प्रोटीन सुपर वर्ग से दूर के रिश्तेदार हैं। दोनों जीव में ऑक्सीजन के परिवहन का ही उद्देश्य पूरा करते हैं। यद्यपि इन दोनों प्रोटीनों में पूरी तरह से भिन्न अमीनो एसिड अनुक्रम हैं, उनकी प्रोटीन संरचनाएं वस्तुतः समान हैं, जो उनके लगभग समान उद्देश्यों और साझा पूर्वज को दर्शाती हैं।[53]
प्रोटीन संरचना की भविष्यवाणी करने की अन्य तकनीकों में प्रोटीन थ्रेडिंग और डे नोवो (स्क्रैच से) भौतिकी-आधारित मॉडलिंग सम्मिलित हैं।
संरचनात्मक जैव सूचना विज्ञान के अन्य कथन में क्यूएसएआर का उपयोग किया जाता है मात्रात्मक संरचना-गतिविधि संबंध मॉडल और प्रोटीओ के मोमेट्रिक मॉडल (पीसीएम) जैसे वर्चुअल स्क्रीनिंग मॉडल के लिए प्रोटीन संरचनाओं का उपयोग सम्मिलित है। इसके अतिरिक्त, प्रोटीन की क्रिस्टल संरचना का उपयोग उदाहरण के लिए लिगैंड-बाइंडिंग अध्ययन और सिलिको उत्परिवर्तन अध्ययन के अनुकरण में किया जा सकता है।
गूगल के डीपमाइंड द्वारा विकसित अल्फ़ाफ़ोल्ड नामक 2021 ध्यान लगा के पढ़ना या सीखना एल्गोरिदम-आधारित सॉफ़्टवेयर, अन्य सभी भविष्यवाणी सॉफ़्टवेयर विधियों से अधिक उत्तम प्रदर्शन करता है[54], और अल्फाफोल्ड प्रोटीन संरचना डेटाबेस में लाखों प्रोटीनों के लिए अनुमानित संरचनाएं जारी की हैं।[55]
नेटवर्क और प्रणालीजीवविज्ञान
नेटवर्क विश्लेषण मेटाबोलिक नेटवर्क या इंटरएक्टोम में उपयोग किया जाता है | प्रोटीन इंटरेक्शन नेटवर्क जैसे जैविक नेटवर्क के अंदर संबंधों को समझने का प्रयास करता है। यद्यपि जैविक नेटवर्क का निर्माण ही प्रकार के अणु या इकाई (जैसे जीन) से किया जा सकता है, नेटवर्क जीव विज्ञान अधिकांशतः अनेकभिन्न -भिन्न डेटा प्रकारों को एकीकृत करने का प्रयास करता है, जैसे कि प्रोटीन, छोटे अणु, जीन अभिव्यक्ति डेटा और अन्य, जो सभी भौतिक रूप से जुड़े हुए हैं , कार्यात्मक रूप से, या दोनों।
प्रणाली बायोलॉजी में इन सेलुलर प्रक्रियाओं के समष्टि कनेक्शनों का विश्लेषण और कल्पना करने के लिए सेल (जीव विज्ञान) उपप्रणालियों (जैसे मेटाबोलिज्म नेटवर्क और एंजाइम जिनमें मेटाबोलिज्म , सिग्नल ट्रांसडक्शन पथ और जीन नियामक नेटवर्क सम्मिलित हैं) के कंप्यूटर सिमुलेशन का उपयोग सम्मिलित है। कृत्रिम जीवन या आभासी विकास सरल (कृत्रिम) जीवन रूपों के कंप्यूटर सिमुलेशन के माध्यम से विकासवादी प्रक्रियाओं को समझने का प्रयास करता है।
आणविक संपर्क नेटवर्क

हजारों त्रि-आयामी प्रोटीन संरचनाएं एक्स-रे क्रिस्टलोग्राफी और प्रोटीन परमाणु चुंबकीय अनुनाद स्पेक्ट्रोस्कोपी (प्रोटीन एनएमआर) द्वारा निर्धारित की गई हैं और संरचनात्मक जैव सूचना विज्ञान में केंद्रीय प्रश्न यह है कि क्या केवल इनके आधार पर संभावित प्रोटीन-प्रोटीन इंटरैक्शन की भविष्यवाणी करना व्यावहारिक है। प्रोटीन-प्रोटीन अंतःक्रिया प्रयोग किए बिना 3डी आकार। प्रोटीन-प्रोटीन डॉकिंग समस्या से निपटने के लिए अनेकतरह के तरीके विकसित किए गए हैं, चूंकि ऐसा लगता है कि इस क्षेत्र में अभी भी बहुत काम किया जाना बाकी है।
क्षेत्र में सामने आने वाली अन्य अंतःक्रियाओं में प्रोटीन-लिगैंड (दवा सहित) और प्रोटीन-पेप्टाइड सम्मिलित हैं। घूमने योग्य बांडों के बारे में परमाणुओं की गति का आणविक गतिशील अनुकरण, इंटरैक्टोम का अध्ययन करने के लिए कम्प्यूटेशनल एल्गोरिदम, जिसे डॉकिंग एल्गोरिदम कहा जाता है, के पीछे मूल सिद्धांत भी है।
अन्य
साहित्य विश्लेषण
प्रकाशित साहित्य की विशाल संख्या व्यक्तियों के लिए प्रत्येक पेपर को पढ़ना लगभग असंभव बना देती है, जिसके परिणामस्वरूप अनुसंधान के उप-क्षेत्र असंबद्ध हो जाते हैं। साहित्य विश्लेषण का उद्देश्य पाठ्य संसाधनों की इस बढ़ती लाइब्रेरी के खनन के लिए कम्प्यूटेशनल और सांख्यिकीय भाषाविज्ञान को नियोजित करना है। उदाहरण के लिए:
- संक्षिप्तीकरण पहचान - जैविक शब्दों के दीर्घ-रूप और संक्षिप्तीकरण की पहचान करें
- नामित-इकाई पहचान - जीन नाम जैसे जैविक शब्दों को पहचानना
- प्रोटीन-प्रोटीन अंतःक्रिया - पाठ से पहचानें कि कौन सा प्रोटीन किस प्रोटीन के साथ अंतःक्रिया करता है
अनुसंधान का क्षेत्र सांख्यिकी और कम्प्यूटेशनल भाषाविज्ञान से आता है।
उच्च-थ्रूपुट छवि विश्लेषण
कम्प्यूटेशनल प्रौद्योगिकियों का उपयोग बड़ी मात्रा में उच्च-सूचना-सामग्री चिकित्सा इमेजिंग के प्रसंस्करण, मात्रा निर्धारण और विश्लेषण को स्वचालित करने के लिए किया जाता है। आधुनिक छवि विश्लेषण प्रणालियाँ पर्यवेक्षक की स्पष्टता, वस्तुनिष्ठता (विज्ञान), या गति में सुधार कर सकती हैं। छवि विश्लेषण निदान और अनुसंधान दोनों के लिए महत्वपूर्ण है। इसके कुछ उदाहरण निम्न हैं:
- उच्च-थ्रूपुट और उच्च-निष्ठा परिमाणीकरण और उप-सेलुलर स्थानीयकरण (उच्च-सामग्री स्क्रीनिंग, साइटोहिस्टोपैथोलॉजी, बायोइमेज सूचना विज्ञान)
- मॉर्फोमेट्रिक्स
- नैदानिक छवि विश्लेषण और विज़ुअलाइज़ेशन
- जीवित जानवरों के सांस लेने वाले फेफड़ों में वास्तविक समय में वायु-प्रवाह पैटर्न का निर्धारण करना
- धमनी की चोट के समय विकास और पुनर्प्राप्ति से वास्तविक समय की इमेजरी में रोड़ा आकार की मात्रा निर्धारित करना
- प्रयोगशाला जानवरों की विस्तारित वीडियो रिकॉर्डिंग से व्यवहार संबंधी अवलोकन करना
- मेटाबोलिज्म गतिविधि निर्धारण के लिए अवरक्त माप
- जीन मैपिंग में क्लोन ओवरलैप का अनुमान लगाना, उदा. सुलस्टन स्कोर करना होता है
उच्च-थ्रूपुट एकल कक्ष डेटा विश्लेषण
कम्प्यूटेशनल तकनीकों का उपयोग उच्च-थ्रूपुट, कम-माप वाले एकल कोशिका डेटा का विश्लेषण करने के लिए किया जाता है, जैसे कि फ़्लो साइटॉमेट्री से प्राप्त किया जाता है। इन विधियों में सामान्यतः उन कोशिकाओं की जनसंख्या का पता लगाना सम्मिलित होता है जो किसी विशेष रोग अवस्था या प्रायोगिक स्थिति के लिए प्रासंगिक होती हैं।
जैव विविधता सूचना विज्ञान
जैव विविधता सूचना विज्ञान जैव विविधता डेटा से , जैसे टैक्सोनोमिक डेटाबेस, या माइक्रोबायोम डेटा के संग्रह और विश्लेषण से संबंधित है। ऐसे विश्लेषणों के उदाहरणों में फ़ाइलोजेनेटिक्स, आला मॉडलिंग, प्रजाति समृद्धि मानचित्रण, डीएनए बारकोडिंग, या प्रजातिवाद पहचान उपकरण सम्मिलित हैं।
ओन्टोलॉजी और डेटा एकीकरण
जैविक ऑन्कोलॉजी नियंत्रित शब्दावली के निर्देशित चक्रीय रेखांकन हैं। वह जैविक अवधारणाओं और विवरणों के लिए श्रेणियां बनाते हैं जिससे कि कंप्यूटर के साथ उनका आसानी से विश्लेषण किया जा सके। जब इस प्रकार वर्गीकृत किया जाता है, तब समग्र और एकीकृत विश्लेषण से अतिरिक्त मूल्य प्राप्त करना संभव है।
ओबीओ फाउंड्री कुछ ऑन्कोलॉजी को मानकीकृत करने का प्रयास था। सबसे व्यापक में से जीन ऑन्टोलॉजी है जो जीन फलन का वर्णन करता है। ऐसी ऑन्कोलॉजी भी हैं जो फेनोटाइप का वर्णन करती हैं।
डेटाबेस
जैव सूचना विज्ञान अनुसंधान और अनुप्रयोगों के लिए डेटाबेस आवश्यक हैं। डीएनए और प्रोटीन अनुक्रम, आणविक संरचना, फेनोटाइप और जैव विविधता सहित अनेक भिन्न -भिन्न प्रकार की जानकारी के लिए डेटाबेस उपस्तिथ हैं। डेटाबेस में अनुभवजन्य डेटा (प्रयोगों से सीधे प्राप्त) और अनुमानित डेटा (उपस्थित डेटा के विश्लेषण से प्राप्त) दोनों सम्मिलित हो सकते हैं। वह किसी विशेष जीव, मार्ग या रुचि के अणु के लिए विशिष्ट हो सकते हैं। वैकल्पिक रूप से, वह अनेकअन्य डेटाबेस से संकलित डेटा को सम्मिलित कर सकते हैं। डेटाबेस के भिन्न -भिन्न प्रारूप, पहुंच तंत्र और सार्वजनिक या निजी हो सकते हैं।
सबसे अधिक उपयोग किए जाने वाले कुछ डेटाबेस नीचे सूचीबद्ध हैं: जैसे
- जैविक अनुक्रम विश्लेषण में प्रयुक्त: जेनबैंक , यूनीप्रोट को उपयोग किया जाता है
- संरचना विश्लेषण में प्रयुक्त: प्रोटीन डेटा बैंक (पीडीबी) को उपयोग किया जाता है
- प्रोटीन परिवार और अनुक्रम रूपांकन खोजने में इंटरप्रो, पीएफएएम को उपयोग किया जाता है:
- अगली पीढ़ी के अनुक्रमण के लिए प्रयुक्त: अनुक्रम पढ़ें पुरालेख का उपयोग किया जाता है
- नेटवर्क विश्लेषण में प्रयुक्त: मेटाबोलिक पाथवे डेटाबेस (केईजीजी, बायोसाइक डेटाबेस संग्रह), इंटरेक्शन विश्लेषण डेटाबेस, कार्यात्मक नेटवर्क का उपयोग किया जाता है
- सिंथेटिक आनुवंशिक परिपथ के डिजाइन में जेनोकैड का उपयोग किया जाता है:
सॉफ़्टवेयर और उपकरण
जैव सूचना विज्ञान सॉफ़्टवेयर की सूची में सरल कमांड-लाइन उपकरण सम्मिलित है,और अधिक समष्टि ग्राफ़िकल प्रोग्राम और स्टैंड अलोन वेब-सेवाएँ भी सम्मिलित हैं। वह जैव सूचना विज्ञान कंपनियों की सूची या सार्वजनिक संस्थानों द्वारा बनाए गए हैं।
ओपन-सोर्स जैव सूचना विज्ञान सॉफ्टवेयर
अनेक मुफ़्त और ओपन-सोर्स सॉफ़्टवेयर उपकरण अस्तित्व में हैं और 1980 के दशक से लगातार बढ़ते रहे हैं।[57] उभरते प्रकार के जैविक रीडआउट के विश्लेषण के लिए नए एल्गोरिदम की निरंतर आवश्यकता, सिलिको प्रयोगों में नवीनता की संभावना और स्वतंत्र रूप से उपलब्ध खुले कोड आधारों के संयोजन ने अनुसंधान समूहों के लिए विज्ञान के वित्त पोषण की परवाह किए बिना दोनों जैव सूचना विज्ञान में योगदान करने के अवसर उत्पन्न किए हैं। ओपन सोर्स उपकरण अधिकांशतः विचारों के इनक्यूबेटर या व्यावसायिक अनुप्रयोगों में समुदाय-समर्थित प्लग-इन (कंप्यूटिंग)|प्लग-इन के रूप में कार्य करते हैं। वह जैव सूचना एकीकरण की चुनौती में सहायता के लिए वास्तविक मानक और साझा ऑब्जेक्ट मॉडल भी प्रदान कर सकते हैं।
ओपन-सोर्स बायोइनफॉरमैटिक्स सॉफ्टवेयर में बायोकंडक्टर, बायोपर्ल, बायोपिथॉन , बायोजावा, बायोजेएस, बायोरूबी, बायोक्लिप्स, ईएमबीओएसएस, .एनईटी बायो, ऑरेंज (सॉफ्टवेयर) इसके बायोइनफॉर्मेटिक्स ऐड-ऑन, अपाचे टवेर्ना, यूजीईएनई और जेनोकैड सम्मिलित हैं।
गैर-लाभकारी ओपन बायोइन्फ़ॉर्मेटिक्स फ़ाउंडेशन[57]और वार्षिक जैव सूचना विज्ञान ओपन सोर्स सम्मेलन ओपन-सोर्स जैव सूचना विज्ञान सॉफ्टवेयर को बढ़ावा देता है।[58]
जैव सूचना विज्ञान में वेब सेवाएँ
क्लाइंट कंप्यूटरों को विश्व के अन्य हिस्सों में सर्वर से एल्गोरिदम, डेटा और कंप्यूटिंग संसाधनों का उपयोग करने की अनुमति देने के लिए सोप - और रेस्ट -आधारित इंटरफेस विकसित किए गए हैं। मुख्य लाभ यह है कि अंतिम उपयोगकर्ताओं को सॉफ़्टवेयर और डेटाबेस रखरखाव ओवरहेड्स से निपटना नहीं पड़ता है।
मूलभूत जैव सूचना विज्ञान सेवाओं को यूरोपीय जैव सूचना विज्ञान संस्थान द्वारा तीन श्रेणियों में वर्गीकृत किया गया है: अनुक्रम संरेखण सॉफ्टवेयर (अनुक्रम खोज सेवाएँ), एकाधिक अनुक्रम संरेखण (एकाधिक अनुक्रम संरेखण), और या अनुक्रम विश्लेषण (जैविक अनुक्रम विश्लेषण)।[59] इन सेवा-अभिमुखता | सेवा-उन्मुख जैव सूचना विज्ञान संसाधनों की उपलब्धता वेब-आधारित जैव सूचना विज्ञान समाधानों की प्रयोज्यता को प्रदर्शित करती है, और एकल वेब-आधारित इंटरफ़ेस के अनुसार सामान्य डेटा प्रारूप के साथ स्टैंड अलोन टूल के संग्रह से लेकर एकीकृत, वितरित और विस्तार योग्य जैव सूचना विज्ञान कार्य प्रवाह प्रबंधन प्रणाली तक होती है। ।
जैव सूचना विज्ञान कार्य प्रवाह प्रबंधन प्रणाली
एक जैव सूचना विज्ञान कार्य प्रवाह प्रबंधन प्रणाली कार्य प्रवाह प्रबंधन प्रणाली का विशेष रूप है जिसे विशेष रूप से जैव सूचना विज्ञान अनुप्रयोग में कम्प्यूटेशनल या डेटा हेरफेर चरणों, या कार्य प्रवाह की श्रृंखला को बनाने और निष्पादित करने के लिए डिज़ाइन किया गया है। ऐसे प्रणालीडिज़ाइन किए गए हैं
- व्यक्तिगत अनुप्रयोग वैज्ञानिकों को अपना स्वयं का कार्य प्रवाह बनाने के लिए उपयोग में आसान वातावरण प्रदान करें,
- वैज्ञानिकों को उनके कार्य प्रवाह निष्पादित करने और वास्तविक समय में उनके परिणाम देखने में सक्षम बनाने के लिए इंटरैक्टिव उपकरण प्रदान करें,
- वैज्ञानिकों के मध्य कार्य प्रवाह को साझा करने और पुन: उपयोग करने की प्रक्रिया को सरल बनाएं, और
- वैज्ञानिकों को कार्य प्रवाह निष्पादन परिणामों की उत्पत्ति और कार्य प्रवाह निर्माण चरणों को ट्रैक करने में सक्षम बनाता है।
यह सेवा देने वाले कुछ प्लेटफ़ॉर्म: गैलेक्सी (कम्प्यूटेशनल जीवविज्ञान) , केप्लर वैज्ञानिक कार्यप्रवाह प्रणाली , अपाचे टवेर्ना, यूजीईएनई, एंडुरिल (कार्य प्रवाह इंजन), उच्च-प्रदर्शन एकीकृत वर्चुअल वातावरण।
बायोकंप्यूट और बायोकंप्यूट ऑब्जेक्ट
2014 में, खाद्य एवं औषधि प्रशासन ने जैव सूचना विज्ञान में प्रतिलिपि प्रस्तुत करने योग्यता पर चर्चा करने के लिए राष्ट्रीय स्वास्थ्य संस्थान बेथेस्डा परिसर में आयोजित सम्मेलन को प्रायोजित किया।[60] अगले तीन वर्षों में, हित धारकों का संघ नियमित रूप से इस बात पर चर्चा करने के लिए मिला कि बायोकंप्यूट प्रतिमान क्या बनेगा।[61] इन हित धारकों में सरकार, उद्योग और शैक्षणिक संस्थाओं के प्रतिनिधि सम्मिलित थे। सत्र के नेताओं ने एफडीए और एनआईएच संस्थानों और केंद्रों की अनेकशाखाओं, ह्यूमन वैरिओम प्रोजेक्ट और चिकित्सा सूचना विज्ञान के लिए यूरोपीय संघ सहित गैर-लाभकारी संस्थाओं और स्टैनफोर्ड विश्वविद्यालय, न्यूयॉर्क जीनोम सेंटर और जॉर्ज वाशिंगटन विश्वविद्यालय सहित अनुसंधान संस्थानों का प्रतिनिधित्व किया।
यह निर्णय लिया गया कि बायोकंप्यूट प्रतिमान डिजिटल 'लैब नोटबुक' के रूप में होगा जो जैव सूचना विज्ञान प्रोटोकॉल की प्रतिलिपि प्रस्तुत करने योग्यता, प्रतिकृति, समीक्षा और पुन: उपयोग की अनुमति देता है। यह समूहों के मध्य विचारों के आदान-प्रदान को आगे बढ़ाते हुए सामान्य कार्मिक प्रवाह के समय अनुसंधान समूह के अंदर अधिक निरंतरता को सक्षम करने के लिए प्रस्तावित किया गया था। यूएस एफडीए ने इस काम को वित्त पोषित किया जिससे कि पाइपलाइनों की जानकारी उनके नियामक कर्मचारियों के लिए अधिक पारदर्शी और सुलभ हो सके।[62]
2016 में, समूह ने बेथेस्डा में एनआईएच को पुनर्गठित किया और बायोकंप्यूट प्रतिमान के उदाहरण,बायोकंप्यूट ऑब्जेक्ट की क्षमता पर चर्चा की। तथा इस कार्य को मानक परीक्षण उपयोग दस्तावेज़ और बायोरेक्सिव पर अपलोड किए गए प्रीप्रिंट पेपर दोनों के रूप में कॉपी किया गया था। बायोकंप्यूट ऑब्जेक्ट जेएसओएन-आकारित रिकॉर्ड को कर्मचारियों, सहयोगियों और नियामकों के मध्य साझा करने की अनुमति देता है।[63][64]
शिक्षा मंच
जैव सूचना विज्ञान को अनेक विश्वविद्यालयों में न केवल व्यक्तिगत मास्टर डिग्री के रूप में पढ़ाया जाता है। जब कि जैव सूचना विज्ञान की कम्प्यूटेशनल प्रकृति इसे शैक्षिक प्रौद्योगिकी कंप्यूटर-सहायता प्राप्त और ऑनलाइन सीखने के लिए उधार देती है।[65][66] जैव सूचना विज्ञान अवधारणाओं और विधियों को सिखाने के लिए डिज़ाइन किए गए सॉफ़्टवेयर प्लेटफ़ॉर्म में रोज़लिंड (शिक्षा मंच) और स्विस इंस्टीट्यूट ऑफ़ बायोइनफ़ॉर्मेटिक्स ट्रेनिंग पोर्टल के माध्यम से प्रस्तुत किए जाने वाले ऑनलाइन पाठ्यक्रम सम्मिलित हैं। कनाडाई जैव सूचना विज्ञान कार्यशालाएँ क्रिएटिव कॉमन्स लाइसेंस के अनुसार अपनी वेबसाइट पर प्रशिक्षण कार्यशालाओं से वीडियो और स्लाइड प्रदान करती है। 4273π प्रोजेक्ट या 4273π प्रोजेक्ट[67] यह मुफ़्त में मुक्त स्रोत शैक्षिक सामग्री भी प्रदान करता है। यह पाठ्यक्रम कम निवेश वाले रास्पबेरी पाई कंप्यूटर पर चलता है और इसका उपयोग वयस्कों और स्कूली विद्यार्थियों को पढ़ाने के लिए किया गया है।[68][69] 4283 को शिक्षाविदों और अनुसंधान कर्मचारियों के संघ द्वारा सक्रिय रूप से विकसित किया गया है, जिन्होंने रास्पबेरी पाई कंप्यूटर और 4283π ऑपरेटिंग प्रणाली का उपयोग करके अनुसंधान स्तर की जैव सूचना विज्ञान चलाया है।[70][71]
बड़े पैमाने पर खुले ऑनलाइन पाठ्यक्रम प्लेटफ़ॉर्म जैव सूचना विज्ञान और संबंधित विषयों में ऑनलाइन प्रमाणन भी प्रदान करते हैं, जिसमें कौरसेरा के जैव सूचना विज्ञान विशेषज्ञता (कैलिफोर्निया विश्वविद्यालय, सैन डिएगो) और जीनोमिक डेटा विज्ञान विशेषज्ञता (जॉन्स हॉपकिन्स विश्वविद्यालय) के साथ-साथ एडएक्स के जीवन विज्ञान एक्ससीरीज़ (हार्वर्ड विश्वविद्यालय) के लिए डेटा विश्लेषण सम्मिलित हैं। ).
सम्मेलन
ऐसे अनेक बड़े सम्मेलन हैं जो जैव सूचना विज्ञान से संबंधित हैं। सबसे उल्लेखनीय उदाहरणों में से कुछ हैं आणविक जीवविज्ञान के लिए इंटेलिजेंट प्रणाली (आईएसएमबी), कम्प्यूटेशनल जीवविज्ञान पर यूरोपीय सम्मेलन (ईसीसीबी), और कम्प्यूटेशनल आणविक जीवविज्ञान में अनुसंधान (आरईसीओएमबी)।
यह भी देखें
- जैव विविधता सूचना विज्ञान
- जैव सूचना विज्ञान कंपनियाँ
- कम्प्यूटेशनल बायोलॉजी
- कम्प्यूटेशनल बायोमॉडलिंग
- कम्प्यूटेशनल जीनोमिक्स
- साइबरजैविक सुरक्षा
- कार्यात्मक जीनोमिक्स
- स्वास्थ्य सूचना
- कम्प्यूटेशनल बायोलॉजी के लिए इंटरनेशनल सोसायटी
- जंपिंग लाइब्रेरी
- जैव सूचना विज्ञान संस्थानों की सूची
- ओपन-सोर्स जैव सूचना विज्ञान सॉफ्टवेयर की सूची
- जैव सूचना विज्ञान पत्रिकाओं की सूची
- मेटाबोलॉमिक्स
- न्यूक्लिक एसिड अनुक्रम
- फाइलोजेनेटिक्स
- प्रोटिओमिक्स
- जीन रोग डेटाबेस
संदर्भ
- ↑ Lesk AM (26 July 2013). "बायोइनफॉरमैटिक्स". Encyclopaedia Britannica. Archived from the original on 14 April 2021. Retrieved 17 April 2017.
- ↑ 2.0 2.1 Sim AY, Minary P, Levitt M (June 2012). "न्यूक्लिक एसिड मॉडलिंग". Current Opinion in Structural Biology. 22 (3): 273–8. doi:10.1016/j.sbi.2012.03.012. PMC 4028509. PMID 22538125.
- ↑ Dawson WK, Maciejczyk M, Jankowska EJ, Bujnicki JM (July 2016). "Coarse-grained modeling of RNA 3D structure". Methods. 103: 138–56. doi:10.1016/j.ymeth.2016.04.026. PMID 27125734.
- ↑ Kmiecik S, Gront D, Kolinski M, Wieteska L, Dawid AE, Kolinski A (July 2016). "मोटे दाने वाले प्रोटीन मॉडल और उनके अनुप्रयोग". Chemical Reviews. 116 (14): 7898–936. doi:10.1021/acs.chemrev.6b00163. PMID 27333362.
- ↑ Wong KC (2016). Computational Biology and Bioinformatics: Gene Regulation. CRC Press/Taylor & Francis Group. ISBN 9781498724975.
- ↑ Joyce AP, Zhang C, Bradley P, Havranek JJ (January 2015). "Structure-based modeling of protein: DNA specificity". Briefings in Functional Genomics. 14 (1): 39–49. doi:10.1093/bfgp/elu044. PMC 4366589. PMID 25414269.
- ↑ Spiga E, Degiacomi MT, Dal Peraro M (2014). "New Strategies for Integrative Dynamic Modeling of Macromolecular Assembly". In Karabencheva-Christova T (ed.). बायोमोलेक्यूलर मॉडलिंग और सिमुलेशन. Advances in Protein Chemistry and Structural Biology. Vol. 96. Academic Press. pp. 77–111. doi:10.1016/bs.apcsb.2014.06.008. ISBN 9780128000137. PMID 25443955.
- ↑ Ciemny M, Kurcinski M, Kamel K, Kolinski A, Alam N, Schueler-Furman O, Kmiecik S (August 2018). "Protein-peptide docking: opportunities and challenges". Drug Discovery Today. 23 (8): 1530–1537. doi:10.1016/j.drudis.2018.05.006. PMID 29733895.
- ↑ Ouzounis, C. A.; Valencia, A. (2003). "Early bioinformatics: the birth of a discipline—a personal view". Bioinformatics. 19 (17): 2176–2190. doi:10.1093/bioinformatics/btg309. PMID 14630646.
- ↑ 10.0 10.1 Hogeweg P (March 2011). Searls DB (ed.). "सैद्धांतिक जीव विज्ञान में जैव सूचना विज्ञान की जड़ें". PLOS Computational Biology. 7 (3): e1002021. Bibcode:2011PLSCB...7E2021H. doi:10.1371/journal.pcbi.1002021. PMC 3068925. PMID 21483479.
- ↑ Hesper B, Hogeweg P (1970). "Bio-informatica: een werkconcept". Kameleon. 1 (6): 28–29.
- ↑ Hesper B, Hogeweg P (2021). "Bio-informatics: a working concept. A translation of "Bio-informatica: een werkconcept" by B. Hesper and P. Hogeweg". arXiv:2111.11832v1 [q-bio.OT].
- ↑ Hogeweg P (1978). "सेलुलर रूपों के विकास का अनुकरण". Simulation. 31 (3): 90–96. doi:10.1177/003754977803100305. S2CID 61206099.
- ↑ Colby B (2022). "संपूर्ण जीनोम अनुक्रमण लागत". Sequencing.com. Archived from the original on 15 March 2022. Retrieved 8 April 2022.
- ↑ Moody G (2004). Digital Code of Life: How Bioinformatics is Revolutionizing Science, Medicine, and Business. ISBN 978-0-471-32788-2.
- ↑ Dayhoff, M.O. (1966) Atlas of protein sequence and structure. National Biomedical Research Foundation, 215 pp.
- ↑ Eck RV, Dayhoff MO (April 1966). "आदिम अमीनो एसिड अनुक्रमों के जीवित अवशेषों के आधार पर फेर्रेडॉक्सिन की संरचना का विकास". Science. 152 (3720): 363–6. Bibcode:1966Sci...152..363E. doi:10.1126/science.152.3720.363. PMID 17775169. S2CID 23208558.
- ↑ Johnson G, Wu TT (January 2000). "Kabat database and its applications: 30 years after the first variability plot". Nucleic Acids Research. 28 (1): 214–8. doi:10.1093/nar/28.1.214. PMC 102431. PMID 10592229.
- ↑ Erickson JW, Altman GG (1979). "A Search for Patterns in the Nucleotide Sequence of the MS2 Genome". Journal of Mathematical Biology. 7 (3): 219–230. doi:10.1007/BF00275725. S2CID 85199492.
- ↑ Shulman MJ, Steinberg CM, Westmoreland N (February 1981). "न्यूक्लियोटाइड अनुक्रमों के कोडिंग फ़ंक्शन को सांख्यिकीय विश्लेषण द्वारा देखा जा सकता है". Journal of Theoretical Biology. 88 (3): 409–20. Bibcode:1981JThBi..88..409S. doi:10.1016/0022-5193(81)90274-5. PMID 6456380.
- ↑ Xiong J (2006). आवश्यक जैव सूचना विज्ञान. Cambridge, United Kingdom: Cambridge University Press. pp. 4. ISBN 978-0-511-16815-4 – via Internet Archive.
- ↑ Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, et al. (February 1977). "Nucleotide sequence of bacteriophage phi X174 DNA". Nature. 265 (5596): 687–95. Bibcode:1977Natur.265..687S. doi:10.1038/265687a0. PMID 870828. S2CID 4206886.
- ↑ Benson DA, Karsch-Mizrachi I, Lipman DJ, Ostell J, Wheeler DL (January 2008). "GenBank". Nucleic Acids Research. 36 (Database issue): D25-30. doi:10.1093/nar/gkm929. PMC 2238942. PMID 18073190.
- ↑ 24.0 24.1 24.2 Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, et al. (July 1995). "हेमोफिलस इन्फ्लुएंजा आरडी का संपूर्ण-जीनोम यादृच्छिक अनुक्रमण और संयोजन". Science. 269 (5223): 496–512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800.
- ↑ Stein, Lincoln (2001). "Genome annotation: from sequence to biology". Nature. 2 (7): 493–503. doi:10.1038/35080529. PMID 11433356. S2CID 12044602.
- ↑ Erdin S, Lisewski AM, Lichtarge O (April 2011). "Protein function prediction: towards integration of similarity metrics". Current Opinion in Structural Biology. 21 (2): 180–8. doi:10.1016/j.sbi.2011.02.001. PMC 3120633. PMID 21353529.
- ↑ Carvajal-Rodríguez A (March 2010). "समय में जीन और जीनोम का अनुकरण". Current Genomics. 11 (1): 58–61. doi:10.2174/138920210790218007. PMC 2851118. PMID 20808525.
- ↑ Brown TA (2002). "Mutation, Repair and Recombination". जीनोम (2nd ed.). Manchester (UK): Oxford.
- ↑ Carter NP, Fiegler H, Piper J (October 2002). "Comparative analysis of comparative genomic hybridization microarray technologies: report of a workshop sponsored by the Wellcome Trust". Cytometry. 49 (2): 43–8. doi:10.1002/cyto.10153. PMID 12357458.
- ↑ Chaudhari NM, Gupta VK, Dutta C (April 2016). "बीपीजीए- एक अल्ट्रा-फास्ट पैन-जीनोम विश्लेषण पाइपलाइन". Scientific Reports. 6: 24373. Bibcode:2016NatSR...624373C. doi:10.1038/srep24373. PMC 4829868. PMID 27071527.
- ↑ Aston KI (May 2014). "Genetic susceptibility to male infertility: news from genome-wide association studies". Andrology. 2 (3): 315–21. doi:10.1111/j.2047-2927.2014.00188.x. PMID 24574159. S2CID 206007180.
- ↑ Véron A, Blein S, Cox DG (2014). "जीनोम-वाइड एसोसिएशन अध्ययन और क्लिनिक: स्तन कैंसर पर ध्यान केंद्रित". Biomarkers in Medicine. 8 (2): 287–96. doi:10.2217/bmm.13.121. PMID 24521025.
- ↑ Tosto G, Reitz C (October 2013). "अल्जाइमर रोग में जीनोम-वाइड एसोसिएशन अध्ययन: एक समीक्षा". Current Neurology and Neuroscience Reports. 13 (10): 381. doi:10.1007/s11910-013-0381-0. PMC 3809844. PMID 23954969.
- ↑ Londin E, Yadav P, Surrey S, Kricka LJ, Fortina P (2013). "Use of linkage analysis, genome-wide association studies, and next-generation sequencing in the identification of disease-causing mutations". फार्माकोजीनोमिक्स. Methods in Molecular Biology. Vol. 1015. pp. 127–46. doi:10.1007/978-1-62703-435-7_8. ISBN 978-1-62703-434-0. PMID 23824853.
- ↑ Hindorff LA, Sethupathy P, Junkins HA, Ramos EM, Mehta JP, Collins FS, Manolio TA (June 2009). "मानव रोगों और लक्षणों के लिए जीनोम-वाइड एसोसिएशन लोकी के संभावित एटियोलॉजिकल और कार्यात्मक निहितार्थ". Proceedings of the National Academy of Sciences of the United States of America. 106 (23): 9362–7. Bibcode:2009PNAS..106.9362H. doi:10.1073/pnas.0903103106. PMC 2687147. PMID 19474294.
- ↑ Hall LO (2010). "Finding the right genes for disease and prognosis prediction". 2010 International Conference on System Science and Engineering. pp. 1–2. doi:10.1109/ICSSE.2010.5551766. ISBN 978-1-4244-6472-2. S2CID 21622726.
{{cite book}}:|journal=ignored (help) - ↑ Manolio, Teri A.; Collins, Francis S.; Cox, Nancy J.; et al. (October 2009). "जटिल रोगों की लुप्त आनुवंशिकता का पता लगाना". Nature. 461 (7265): 747–753. Bibcode:2009Natur.461..747M. doi:10.1038/nature08494. PMC 2831613. PMID 19812666.
- ↑ Wainschtein, Pierrick; Jain, Deepti; Zheng, Zhili; et al. (March 2022). "संपूर्ण-जीनोम अनुक्रम डेटा से जटिल लक्षण आनुवंशिकता में दुर्लभ वेरिएंट के योगदान का आकलन करना". Nature Genetics. 54 (3): 263–273. doi:10.1038/s41588-021-00997-7. PMC 9119698. PMID 35256806.
- ↑ Taliun, Daniel; Harris, Daniel N.; Kessler, Michael D.; et al. (February 2021). "Sequencing of 53,831 diverse genomes from the NHLBI TOPMed Program". Nature. 590 (7845): 290–299. Bibcode:2021Natur.590..290T. doi:10.1038/s41586-021-03205-y. PMC 7875770. PMID 33568819.
- ↑ Li, Xihao; Li, Zilin; Zhou, Hufeng; et al. (September 2020). "सिलिको कार्यात्मक एनोटेशन में मल्टीपल का गतिशील समावेश बड़े पैमाने पर पूरे-जीनोम अनुक्रमण अध्ययनों के दुर्लभ वैरिएंट एसोसिएशन विश्लेषण को सशक्त बनाता है". Nature Genetics. 52 (9): 969–983. doi:10.1038/s41588-020-0676-4. PMC 7483769. PMID 32839606.
- ↑ Li, Zilin; Li, Xihao; Zhou, Hufeng; et al. (December 2022). "बड़े पैमाने पर संपूर्ण-जीनोम अनुक्रमण अध्ययनों के गैर-कोडिंग दुर्लभ-संस्करण संघों का पता लगाने के लिए एक रूपरेखा". Nature Methods. 19 (12): 1599–1611. doi:10.1038/s41592-022-01640-x. PMC 10008172. PMID 36303018. S2CID 243873361.
- ↑ "STAARpipeline: an all-in-one rare-variant tool for biobank-scale whole-genome sequencing data". Nature Methods. 19 (12): 1532–1533. December 2022. doi:10.1038/s41592-022-01641-w. PMID 36316564. S2CID 253246835.
- ↑ Li, Xihao; Quick, Corbin; Zhou, Hufeng; Gaynor, Sheila M.; Liu, Yaowu; Chen, Han; Selvaraj, Margaret Sunitha; Sun, Ryan; Dey, Rounak; Arnett, Donna K.; Bielak, Lawrence F.; Bis, Joshua C.; Blangero, John; Boerwinkle, Eric; Bowden, Donald W.; Brody, Jennifer A.; Cade, Brian E.; Correa, Adolfo; Cupples, L. Adrienne; Curran, Joanne E.; de Vries, Paul S.; Duggirala, Ravindranath; Freedman, Barry I.; Göring, Harald H. H.; Guo, Xiuqing; Haessler, Jeffrey; Kalyani, Rita R.; Kooperberg, Charles; Kral, Brian G.; Lange, Leslie A.; Manichaikul, Ani; Martin, Lisa W.; McGarvey, Stephen T.; Mitchell, Braxton D.; Montasser, May E.; Morrison, Alanna C.; Naseri, Take; O’Connell, Jeffrey R.; Palmer, Nicholette D.; Peyser, Patricia A.; Psaty, Bruce M.; Raffield, Laura M.; Redline, Susan; Reiner, Alexander P.; Reupena, Muagututi’a Sefuiva; Rice, Kenneth M.; Rich, Stephen S.; Sitlani, Colleen M.; Smith, Jennifer A.; Taylor, Kent D.; Vasan, Ramachandran S.; Willer, Cristen J.; Wilson, James G.; Yanek, Lisa R.; Zhao, Wei; NHLBI Trans-Omics for Precision Medicine (TOPMed) Consortium; TOPMed Lipids Working Group; Rotter, Jerome I.; Natarajan, Pradeep; Peloso, Gina M.; Li, Zilin; Lin, Xihong (January 2023). "बड़े संपूर्ण जीनोम अनुक्रमण अध्ययनों में दुर्लभ प्रकार के संघों का शक्तिशाली, स्केलेबल और संसाधन-कुशल मेटा-विश्लेषण". Nature Genetics. 55 (1): 154–164. doi:10.1038/s41588-022-01225-6. PMC 10084891. PMID 36564505. S2CID 255084231.
- ↑ Vazquez M, de la Torre V, Valencia A (2012-12-27). "Chapter 14: Cancer genome analysis". PLOS Computational Biology. 8 (12): e1002824. Bibcode:2012PLSCB...8E2824V. doi:10.1371/journal.pcbi.1002824. PMC 3531315. PMID 23300415.
- ↑ Hye-Jung EC, Jaswinder K, Martin K, Samuel AA, Marco AM (2014). "Second-Generation Sequencing for Cancer Genome Analysis". In Dellaire G, Berman JN, Arceci RJ (eds.). कैंसर जीनोमिक्स. Boston (US): Academic Press. pp. 13–30. doi:10.1016/B978-0-12-396967-5.00002-5. ISBN 9780123969675.
- ↑ Grau J, Ben-Gal I, Posch S, Grosse I (July 2006). "VOMBAT: prediction of transcription factor binding sites using variable order Bayesian trees". Nucleic Acids Research. 34 (Web Server issue): W529-33. doi:10.1093/nar/gkl212. PMC 1538886. PMID 16845064.
- ↑ "मानव प्रोटीन एटलस". www.proteinatlas.org. Archived from the original on 4 March 2020. Retrieved 2017-10-02.
- ↑ "मानव कोशिका". www.proteinatlas.org. Archived from the original on 2 October 2017. Retrieved 2017-10-02.
- ↑ Thul PJ, Åkesson L, Wiking M, Mahdessian D, Geladaki A, Ait Blal H, et al. (May 2017). "मानव प्रोटीओम का एक उपकोशिकीय मानचित्र". Science. 356 (6340): eaal3321. doi:10.1126/science.aal3321. PMID 28495876. S2CID 10744558.
- ↑ Ay F, Noble WS (September 2015). "Analysis methods for studying the 3D architecture of the genome". Genome Biology. 16 (1): 183. doi:10.1186/s13059-015-0745-7. PMC 4556012. PMID 26328929.
- ↑ Kryshtafovych, A.; Schwede, T.; Topf, M.; Fidelis, K.; Moult, J. (2019). "Critical Assessment of Methods of Protein Structure Prediction (CASP) – Round XIII". Proteins. 87 (12): 1011–1020. doi:10.1002/prot.25823. PMC 6927249. PMID 31589781.
- ↑ "Home - CASP14". predictioncenter.org. Archived from the original on 30 January 2023. Retrieved 2023-06-12.
- ↑ Hoy JA, Robinson H, Trent JT, Kakar S, Smagghe BJ, Hargrove MS (August 2007). "Plant hemoglobins: a molecular fossil record for the evolution of oxygen transport". Journal of Molecular Biology. 371 (1): 168–79. doi:10.1016/j.jmb.2007.05.029. PMID 17560601.
- ↑ Jumper, John; Evans, Richard; Pritzel, Alexander; Green, Tim; Figurnov, Michael; Ronneberger, Olaf; Tunyasuvunakool, Kathryn; Bates, Russ; Žídek, Augustin; Potapenko, Anna; Bridgland, Alex; Meyer, Clemens; Kohl, Simon A. A.; Ballard, Andrew J.; Cowie, Andrew (August 2021). "अल्फाफोल्ड के साथ अत्यधिक सटीक प्रोटीन संरचना भविष्यवाणी". Nature (in English). 596 (7873): 583–589. Bibcode:2021Natur.596..583J. doi:10.1038/s41586-021-03819-2. ISSN 1476-4687. PMC 8371605. PMID 34265844.
- ↑ "अल्फाफोल्ड प्रोटीन संरचना डेटाबेस". alphafold.ebi.ac.uk. Archived from the original on 24 July 2021. Retrieved 2022-10-10.
- ↑ Titz B, Rajagopala SV, Goll J, Häuser R, McKevitt MT, Palzkill T, Uetz P (May 2008). Hall N (ed.). "ट्रेपोनिमा पैलिडम का बाइनरी प्रोटीन इंटरैक्टोम - सिफलिस स्पाइरोकेट". PLOS ONE. 3 (5): e2292. Bibcode:2008PLoSO...3.2292T. doi:10.1371/journal.pone.0002292. PMC 2386257. PMID 18509523.
- ↑ 57.0 57.1 "Open Bioinformatics Foundation: About us". Official website. Open Bioinformatics Foundation. Archived from the original on 12 May 2011. Retrieved 10 May 2011.
- ↑ "Open Bioinformatics Foundation: BOSC". Official website. Open Bioinformatics Foundation. Archived from the original on 18 July 2011. Retrieved 10 May 2011.
- ↑ Nisbet R, Elder IV J, Miner G (2009). "Bioinformatics". सांख्यिकीय विश्लेषण और डेटा खनन अनुप्रयोगों की पुस्तिका. Academic Press. p. 328. ISBN 978-0080912035.
- ↑ Office of the Commissioner. "Advancing Regulatory Science – Sept. 24–25, 2014 Public Workshop: Next Generation Sequencing Standards". www.fda.gov (in English). Archived from the original on 14 November 2017. Retrieved 2017-11-30.
- ↑ Simonyan V, Goecks J, Mazumder R (2017). "बायोकंप्यूट ऑब्जेक्ट्स-बायोमेडिकल वैज्ञानिक संगणनाओं के मूल्यांकन और सत्यापन की दिशा में एक कदम". PDA Journal of Pharmaceutical Science and Technology. 71 (2): 136–146. doi:10.5731/pdajpst.2016.006734. PMC 5510742. PMID 27974626.
- ↑ Office of the Commissioner. "Advancing Regulatory Science – Community-based development of HTS standards for validating data and computation and encouraging interoperability". www.fda.gov (in English). Archived from the original on 26 January 2018. Retrieved 2017-11-30.
- ↑ Alterovitz G, Dean D, Goble C, Crusoe MR, Soiland-Reyes S, Bell A, et al. (December 2018). "एचटीएस उत्पत्ति, विश्लेषण और परिणामों के मानक संचार के माध्यम से सटीक चिकित्सा को सक्षम करना". PLOS Biology. 16 (12): e3000099. doi:10.1371/journal.pbio.3000099. PMC 6338479. PMID 30596645.
- ↑ BioCompute Object (BCO) project is a collaborative and community-driven framework to standardize HTS computational data. 1. BCO Specification Document: user manual for understanding and creating B., biocompute-objects, 2017-09-03, archived from the original on 27 June 2018, retrieved 30 November 2017
- ↑ Campbell, A. Malcolm (2003-06-01). "जीनोमिक्स, प्रोटिओमिक्स और जैव सूचना विज्ञान शिक्षण के लिए सार्वजनिक पहुंच". Cell Biology Education. 2 (2): 98–111. doi:10.1187/cbe.03-02-0007. PMC 162192. PMID 12888845.
- ↑ Arenas, Miguel (September 2021). "General considerations for online teaching practices in bioinformatics in the time of COVID ‐19". Biochemistry and Molecular Biology Education (in English). 49 (5): 683–684. doi:10.1002/bmb.21558. ISSN 1470-8175. PMC 8426940. PMID 34231941.
- ↑ Barker D, Ferrier DE, Holland PW, Mitchell JB, Plaisier H, Ritchie MG, Smart SD (August 2013). "4273π: bioinformatics education on low cost ARM hardware". BMC Bioinformatics. 13: 522. doi:10.1186/1471-2105-14-243. PMC 3751261. PMID 23937194.
- ↑ Barker D, Alderson RG, McDonagh JL, Plaisier H, Comrie MM, Duncan L, et al. (2015). "University-level practical activities in bioinformatics benefit voluntary groups of pupils in the last 2 years of school". International Journal of STEM Education. 2 (17). doi:10.1186/s40594-015-0030-z. S2CID 256396656.
- ↑ McDonagh JL, Barker D, Alderson RG (2016). "कम्प्यूटेशनल विज्ञान को जनता के सामने लाना". SpringerPlus. 5 (259): 259. doi:10.1186/s40064-016-1856-7. PMC 4775721. PMID 27006868.
- ↑ Robson JF, Barker D (October 2015). "रास्पबेरी पाई कंप्यूटर का उपयोग करके क्लैमाइडिया ट्रैकोमैटिस और प्रोटोक्लामाइडिया अमीबोफिला की प्रोटीन-कोडिंग जीन सामग्री की तुलना". BMC Research Notes. 8 (561): 561. doi:10.1186/s13104-015-1476-2. PMC 4604092. PMID 26462790.
- ↑ Wreggelsworth KM, Barker D (October 2015). "दो हरे सल्फर बैक्टीरिया, क्लोरोबियम टेपिडम टीएलएस और पेलोडिक्टन फियोक्लैथ्रेटिफोर्म बीयू-1 के प्रोटीन-कोडिंग जीनोम की तुलना". BMC Research Notes. 8 (565): 565. doi:10.1186/s13104-015-1535-8. PMC 4606965. PMID 26467441.
अग्रिम पठन
- Sehgal et al. : Structural, phylogenetic and docking studies of D-amino acid oxidase activator(DAOA ), a candidate schizophrenia gene. Theoretical Biology and Medical Modelling 2013 10 :3.
- Achuthsankar S Nair Computational Biology & Bioinformatics – A gentle Overview Archived 16 December 2008 at the Wayback Machine, Communications of Computer Society of India, January 2007
- Aluru, Srinivas, ed. Handbook of Computational Molecular Biology. Chapman & Hall/Crc, 2006. ISBN 1-58488-406-1 (Chapman & Hall/Crc Computer and Information Science Series)
- Baldi, P and Brunak, S, Bioinformatics: The Machine Learning Approach, 2nd edition. MIT Press, 2001. ISBN 0-262-02506-X
- Barnes, M.R. and Gray, I.C., eds., Bioinformatics for Geneticists, first edition. Wiley, 2003. ISBN 0-470-84394-2
- Baxevanis, A.D. and Ouellette, B.F.F., eds., Bioinformatics: A Practical Guide to the Analysis of Genes and Proteins, third edition. Wiley, 2005. ISBN 0-471-47878-4
- Baxevanis, A.D., Petsko, G.A., Stein, L.D., and Stormo, G.D., eds., Current Protocols in Bioinformatics. Wiley, 2007. ISBN 0-471-25093-7
- Cristianini, N. and Hahn, M. Introduction to Computational Genomics Archived 4 January 2009 at the Wayback Machine, Cambridge University Press, 2006. (ISBN 9780521671910 |ISBN 0-521-67191-4)
- Durbin, R., S. Eddy, A. Krogh and G. Mitchison, Biological sequence analysis. Cambridge University Press, 1998. ISBN 0-521-62971-3
- Gilbert D (September 2004). "Bioinformatics software resources". Briefings in Bioinformatics. 5 (3): 300–4. doi:10.1093/bib/5.3.300. PMID 15383216.
- Keedwell, E., Intelligent Bioinformatics: The Application of Artificial Intelligence Techniques to Bioinformatics Problems. Wiley, 2005. ISBN 0-470-02175-6
- Kohane, et al. Microarrays for an Integrative Genomics. The MIT Press, 2002. ISBN 0-262-11271-X
- Lund, O. et al. Immunological Bioinformatics. The MIT Press, 2005. ISBN 0-262-12280-4
- Pachter, Lior and Sturmfels, Bernd. "Algebraic Statistics for Computational Biology" Cambridge University Press, 2005. ISBN 0-521-85700-7
- Pevzner, Pavel A. Computational Molecular Biology: An Algorithmic Approach The MIT Press, 2000. ISBN 0-262-16197-4
- Soinov, L. Bioinformatics and Pattern Recognition Come Together Archived 10 May 2013 at the Wayback Machine Journal of Pattern Recognition Research (JPRR Archived 8 September 2008 at the Wayback Machine), Vol 1 (1) 2006 p. 37–41
- Stevens, Hallam, Life Out of Sequence: A Data-Driven History of Bioinformatics, Chicago: The University of Chicago Press, 2013, ISBN 9780226080208
- Tisdall, James. "Beginning Perl for Bioinformatics" O'Reilly, 2001. ISBN 0-596-00080-4
- Catalyzing Inquiry at the Interface of Computing and Biology (2005) CSTB report Archived 28 January 2007 at the Wayback Machine
- Calculating the Secrets of Life: Contributions of the Mathematical Sciences and computing to Molecular Biology (1995) Archived 6 July 2008 at the Wayback Machine
- Foundations of Computational and Systems Biology MIT Course
- Computational Biology: Genomes, Networks, Evolution Free MIT Course Archived 8 April 2013 at the Wayback Machine
बाहरी संबंध
| Library resources about बायोइनफॉरमैटिक्स |
 The dictionary definition of bioinformatics at Wiktionary
The dictionary definition of bioinformatics at Wiktionary Learning materials related to बायोइनफॉरमैटिक्स at Wikiversity
Learning materials related to बायोइनफॉरमैटिक्स at Wikiversity Media related to बायोइनफॉरमैटिक्स at Wikimedia Commons
Media related to बायोइनफॉरमैटिक्स at Wikimedia Commons- Bioinformatics Resource Portal (SIB)